近期,中国农业科学院蔬菜花卉研究所王晓武团队在Nucleic Acids Research在线发表题为BRAD V3.0: an upgraded Brassicaceae database的研究论文。报道了第三版十字花科数据库(BRAD V3.0)的重建升级。该数据库整合了目前已发表的十字花科物种的基因组、转录组等组学数据资源,已成为最重要的十字花科植物数据库之一,对十字花科植物组学数据资源的利用和分子设计育种的研究具有重要的价值。
十字花科植物包含模式植物拟南芥,以及白菜、甘蓝、油菜等重要的蔬菜和油料作物,具有非常重要的科研和经济价值。其中许多重要物种的大量组学数据(包括基因组、转录组和变异组等)已陆续被研究和报道。王晓武团队于2011年率先主导完成白菜基因组的测序及组装,并先后参与完成了甘蓝,芥菜、油菜的基因组的测序和组装。该团队基于白菜和拟南芥的基因组数据资源于2011年建立了十字花科芸苔属作物基因组数据库网站Brassica database(BRAD, http://brassicadb.org),已成为十字花科基因组研究的重要门户数据库。在此基础上,2015年该数据库整合了13个十字花科物种的基因组数据及共线性关系的信息,BRAD数据库网站也升级到了2.0版本(Database-Oxford)。该数据库极大地促进了国内外芸薹属物种的研究。随着测序技术的发展,越来越多的十字花科基因组、转录组等组学数据被公布,BRAD数据库面临不断地更新、升级的需求。
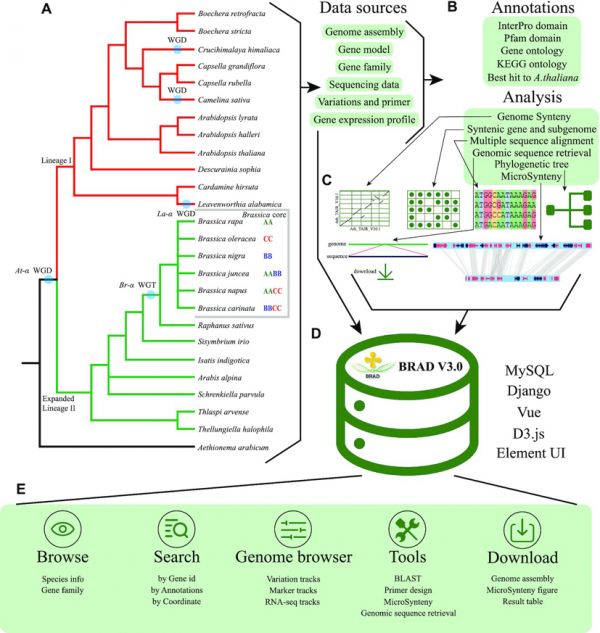
本研究通过重建BRAD数据库,累计收录整合了36个十字花科物种基因组及308份样品的转录组数据和3个物种(白菜、甘蓝和油菜)群体变异数据,并搭建了共线性分析等重要工具,升级为 BRAD V3.0 数据库,主要提供十字花科基因组资源的访问、下载、在线分析等功能。其主要特点在于提供十字花科物种间基因共线性关系,极大地方便了十字花科作物研究人员利用拟南芥基因功能信息开展深入研究。另外,该数据库还新增了微共线性、共线性基因、系统发育树构建、变异数据检索、转录组数据检索、基因序列比对和引物设计等特色功能。简单且用户友好的网络界面, 使得研究人员和育种工作者更加容易地进行数据访问、查询和分析。该数据库将持续更新十字花科物种组学数据,将极大提升十字花科物种数据利用和深度挖掘能力,为十字花科作物功能基因组研究和分子设计育种提供信息平台和数据支撑。
王晓武团队的博士研究生陈海旭为本论文的第一作者,王晓武研究员为本论文的通讯作者。该研究得到了国家重点科研项目、国家自然科学基金以及农业科技创新项目等项目资助。
原文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab1057/6424756
(宣传信息员 林润茂)
相关附件
NAR__Figure1.zip综合新闻
 10/31 2024 蔬菜花卉所6个甘蓝品种入选全国十大品种
10/31 2024 蔬菜花卉所6个甘蓝品种入选全国十大品种  10/31 2024 蔬菜花卉所第五党支部开展“弘扬爱国主义精神,凝聚团结奋进力量”主题党日活动
10/31 2024 蔬菜花卉所第五党支部开展“弘扬爱国主义精神,凝聚团结奋进力量”主题党日活动 
