PlantCFG: 植物开花基因数据库

导语

PlantCFG致力于收集、组织、展示和比较植物基因组中的开花基因。通过PlantCFG,用户可以有效地获得不同物种的候选开花基因信息,而无需在不同的数据库中来回导航。

要点

植物发育从营养阶段到生殖阶段的过渡,即花的过渡,在植物的生命周期中发挥着重要作用。开花是建立作物产量的一个脆弱但关键的阶段,因此,这一时期的适当时机对获得最佳产量至关重要。因此,开花时间的分析是植物分子遗传学、进化生物学、生态学和作物育种等领域的热门话题。近年来,在理解不同植物的开花调控方面取得了重大进展,特别是在模式植物拟南芥中。克隆了一些与开花有关的关键基因,并揭示了相关的分子机制。不同植物的开花时间差异很大,可能受到各种环境因素的影响。全面了解多种植物开花基因的功能和系统比较对于阐明作物驯化和适应至关重要。然而,现有的数据库只专注于研究少数模式植物的开花基因,如拟南芥。例如,FLOR-ID是一个专门的开花基因数据库,提供了有关拟南芥开花时间控制的基因网络的详细信息(最后更新于2015年9月23日)。其他植物物种,如水稻、小麦、大豆和甘蓝型油菜,缺乏系统的数据库。从多种植物和不同的数据库中搜索基因既费力又耗时。此外,开花基因不能在项目之间直接进行比较,因为它们来自不同的生物信息学管道。为了提高对开花时间变化的理解,迫切需要一个全面的开花基因数据库,使用户能够比较不同物种的开花基因。
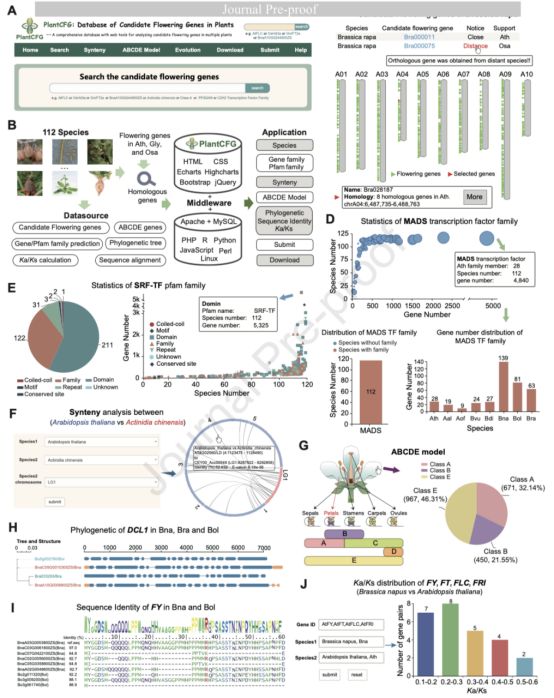 Figure 1 PlantCFG数据库总览
Figure 1 PlantCFG数据库总览
因此,作者开发了一个基于web的集成平台——植物候选开花基因数据库(PlantCFG,https://yanglab.hzau.edu.cn/PlantCFG)(图1A)。共从EnsemblPlants中收集了112个基因组(https://plants.ensembl.org)和以前的出版物。进行了仔细的文献调查,分别在拟南芥、大豆和水稻中筛选出459、71和112个已报道的开花基因。基于已报道的开花基因,通过结合序列相似性和基于保守结构域的方法鉴定了其他物种的候选开花基因。PlantCFG包含从10个植物科获得的82,104个候选开花基因。两种方法共支持52,647个(64.1%)候选开花基因,12,353个由至少两个物种支持。PlantCFG旨在收集、组织、展示和比较多种植物的开花基因,包括以下内容:(i)查询112种植物的候选开花基因的基本信息;(ii)探索“ABCDE”模型中涉及的基因的功能;(iii)对不同物种的候选开花基因进行共线性关系、系统发育分析、序列同一性和Ka/Ks分析;以及(iv)浏览、搜索和批量下载候选开花基因列表和序列(图1B)。PlantCFG由四个主要模块组成:(i)搜索,(ii)同源性,(iii)ABCDE模型和(iv)进化。主页允许用户快速访问四个主要模块和外部链接,包括FLOR-ID、TAIR、EnsemblPlants、InterPro和UniProt。在“搜索”模块中,用户可以通过五个维度检索候选开花基因:(i)物种名称;(ii)已报道的开花基因;(iii)基因家族;(iv)Pfam家族和(v)序列。“按物种搜索”页面显示了112种植物的物种树。相应的属和候选开花基因编号附在每个节点上。用户可以通过单击物种名称或图像按钮,查看每个物种的详细描述,以及候选开花基因的全局统计、功能注释、分布和基因家族分类。可以通过点击染色体或支架来查看候选开花基因的基因组位置信息(图1C)。大多数微调开花时间的机制在不同的植物群中独立进化。对于不属于Brassicaceae、Poceae和Fabeae的物种(使用拟南芥、水稻和大豆作为模式物种),在数据库中实现了“警告”弹出窗口(图1C)。此外,通过点击“变异”和“表达”按钮,可以访问17个物种的候选开花基因的突变和表达信息。如果用户在“按报告的开花基因搜索”中输入有效的基因符号,将显示一个链接,从而更容易获得候选开花基因的信息。将112个品种的候选开花基因分为62个基因家族和372个蛋白质类。最大的基因和Pfam家族是MADS转录因子家族(图1d)和SRF型转录因子(Pfam ID:PF00319)(图1E)。在“按基因家族搜索”和“按Pfam家族搜索”页面上,通过选择基因或Pfam家族的名称,可以获得特定基因或Pfam家族在112个物种中的统计、分布、比较和序列信息(图1D,E)。为了进一步方便用户进行跨物种比较,研究者提供了一个“Synteny”模块来分析不同物种之间候选开花基因的共线性关系,用户可以查看同源基因对的信息(图1F)。与花器官发生相关的基因可分为5组:A、B、C、D和E。为了探索这些基因对花器官发育的调控机制,设计了“ABCDE”模块。总体而言,在ABCDE模型的5类112种植物中鉴定了3227个基因(图1G)。提供了简单而优雅的花器官SVG,用户可以通过点击任何元素来访问调节不同花器官的A、B、C、D和E类同源基因的分级组合(图1G)。“进化”模块由系统发育、序列识别和Ka/Ks分析工具组成。“系统发育”页面提供了候选开花基因系统发育关系的跨物种比较,用户可以访问开花基因的直系同源和旁系同源的完整视图(图1H)。为了获得基因家族中共享的同源基因,在系统发育模块中提供了一种“家族”模式来对基因家族进行系统发育分析。在“序列识别”工具中,用户可以查看每个基因的详细信息,并使用多序列比对查看器进行基因共线性分析。序列logo位于图形顶部,其中字母的高度对应于相应氨基酸的保留量(图1I)。Ka/Ks比值用于诊断序列进化的形式。“Ka/Ks”模块为用户提供了多个动态图表,用于可视化和调查Ka/Ks分布(图1J)。该模块将促进对多种植物开花基因进化的研究。在“下载”模块中,用户可以批量下载FASTA格式的候选开花基因列表以及蛋白质、CDS和基因序列。该模块便于用户高效地执行序列分析。此外,研究者还开发了“提交”模块,用户可以轻松添加新报告的开花基因。为了方便用户更好地理解模块,添加了“关于”页面来搜索、合成、进化和提交模块,并提供了每个模块的详细描述。

小结

PlantCFG致力于收集、组织、展示和比较植物基因组中的开花基因。通过PlantCFG,用户可以有效地获得不同物种的候选开花基因信息,而无需在不同的数据库中来回导航。“Synteny”、“Phyloggenetic”、“Sequence identity”和“Ka/Ks”模块中提供的候选开花基因的跨物种比较功能将有助于阐明作物驯化和适应的分子基础。“系统发育”模块中的基因家族系统发育分析功能将有助于基因家族中共享同源基因的鉴定,并验证已鉴定同源基因对的准确性。用户可以使用“提交”模块提交开花基因,新的开花基因将被审查并定期更新到数据库中。未来,PlantCFG将通过整合更多的基因组、植物和更用户友好的功能来继续发展。这些努力将使PlantCFG成为植物开花基因比较和进化研究的更有价值的资源。
文献来源
PlantCFG: A comprehensive database with web tools for analyzing candidate flowering genes in multiple plants,
PLANT COMMUNICATIONS,2023
相关知识
华中农大和扬州大学联合发布植物开花基因数据库,促进跨物种开花基因比较研究
植物科学常用数据库大全
认真阅读材料一二回答问题.材料一:2012年1
基因工程自定义蜜蜂属性方法
开花植物
园艺中心
洋甘菊,花,昆虫,植物,开花植物,开花植物,脆弱性,野生动物
绿色开花植物
春天开花的植物有哪些 哪些植物春天开花
开花植物施什么肥 开花植物施哪些肥
网址: PlantCFG: 植物开花基因数据库 https://m.huajiangbk.com/newsview626850.html

 PlantCFG致力于收集、组织、展示和比较植物基因组中的开花基因。通过PlantCFG,用户可以有效地获得不同物种的候选开花基因信息,而无需在不同的数据库中来回导航。
PlantCFG致力于收集、组织、展示和比较植物基因组中的开花基因。通过PlantCFG,用户可以有效地获得不同物种的候选开花基因信息,而无需在不同的数据库中来回导航。
 植物发育从营养阶段到生殖阶段的过渡,即花的过渡,在植物的生命周期中发挥着重要作用。开花是建立作物产量的一个脆弱但关键的阶段,因此,这一时期的适当时机对获得最佳产量至关重要。因此,开花时间的分析是植物分子遗传学、进化生物学、生态学和作物育种等领域的热门话题。近年来,在理解不同植物的开花调控方面取得了重大进展,特别是在模式植物拟南芥中。克隆了一些与开花有关的关键基因,并揭示了相关的分子机制。不同植物的开花时间差异很大,可能受到各种环境因素的影响。全面了解多种植物开花基因的功能和系统比较对于阐明作物驯化和适应至关重要。然而,现有的数据库只专注于研究少数模式植物的开花基因,如拟南芥。例如,FLOR-ID是一个专门的开花基因数据库,提供了有关拟南芥开花时间控制的基因网络的详细信息(最后更新于2015年9月23日)。其他植物物种,如水稻、小麦、大豆和甘蓝型油菜,缺乏系统的数据库。从多种植物和不同的数据库中搜索基因既费力又耗时。此外,开花基因不能在项目之间直接进行比较,因为它们来自不同的生物信息学管道。为了提高对开花时间变化的理解,迫切需要一个全面的开花基因数据库,使用户能够比较不同物种的开花基因。
植物发育从营养阶段到生殖阶段的过渡,即花的过渡,在植物的生命周期中发挥着重要作用。开花是建立作物产量的一个脆弱但关键的阶段,因此,这一时期的适当时机对获得最佳产量至关重要。因此,开花时间的分析是植物分子遗传学、进化生物学、生态学和作物育种等领域的热门话题。近年来,在理解不同植物的开花调控方面取得了重大进展,特别是在模式植物拟南芥中。克隆了一些与开花有关的关键基因,并揭示了相关的分子机制。不同植物的开花时间差异很大,可能受到各种环境因素的影响。全面了解多种植物开花基因的功能和系统比较对于阐明作物驯化和适应至关重要。然而,现有的数据库只专注于研究少数模式植物的开花基因,如拟南芥。例如,FLOR-ID是一个专门的开花基因数据库,提供了有关拟南芥开花时间控制的基因网络的详细信息(最后更新于2015年9月23日)。其他植物物种,如水稻、小麦、大豆和甘蓝型油菜,缺乏系统的数据库。从多种植物和不同的数据库中搜索基因既费力又耗时。此外,开花基因不能在项目之间直接进行比较,因为它们来自不同的生物信息学管道。为了提高对开花时间变化的理解,迫切需要一个全面的开花基因数据库,使用户能够比较不同物种的开花基因。 Figure 1 PlantCFG数据库总览
Figure 1 PlantCFG数据库总览
 PlantCFG致力于收集、组织、展示和比较植物基因组中的开花基因。通过PlantCFG,用户可以有效地获得不同物种的候选开花基因信息,而无需在不同的数据库中来回导航。“Synteny”、“Phyloggenetic”、“Sequence identity”和“Ka/Ks”模块中提供的候选开花基因的跨物种比较功能将有助于阐明作物驯化和适应的分子基础。“系统发育”模块中的基因家族系统发育分析功能将有助于基因家族中共享同源基因的鉴定,并验证已鉴定同源基因对的准确性。用户可以使用“提交”模块提交开花基因,新的开花基因将被审查并定期更新到数据库中。未来,PlantCFG将通过整合更多的基因组、植物和更用户友好的功能来继续发展。这些努力将使PlantCFG成为植物开花基因比较和进化研究的更有价值的资源。
PlantCFG致力于收集、组织、展示和比较植物基因组中的开花基因。通过PlantCFG,用户可以有效地获得不同物种的候选开花基因信息,而无需在不同的数据库中来回导航。“Synteny”、“Phyloggenetic”、“Sequence identity”和“Ka/Ks”模块中提供的候选开花基因的跨物种比较功能将有助于阐明作物驯化和适应的分子基础。“系统发育”模块中的基因家族系统发育分析功能将有助于基因家族中共享同源基因的鉴定,并验证已鉴定同源基因对的准确性。用户可以使用“提交”模块提交开花基因,新的开花基因将被审查并定期更新到数据库中。未来,PlantCFG将通过整合更多的基因组、植物和更用户友好的功能来继续发展。这些努力将使PlantCFG成为植物开花基因比较和进化研究的更有价值的资源。