诺贝尔化学奖也花落AI领域!AlphaFold开发者等人获奖

▎药明康德内容团队编辑
就在人工智能学者出人意料地摘得2024年诺贝尔物理学奖之后,北京时间10月9日下午,诺贝尔基金会宣布将今年的诺贝尔化学奖颁发给了三位研究领域与人工智能相关的科学家。
其中,被誉为“AlphaFold之父”的谷歌DeepMind公司Demis Hassabis博士和John Jumper博士因为蛋白质结构预测获奖,而华盛顿大学的David Baker教授因计算蛋白设计荣获殊荣。他们开发的AI解决方案成功解决了50年来蛋白质结构预测领域的重大挑战,加速了生物医药领域的科学发现。

▲David Baker教授、Demis Hassabis博士与John Jumper博士(图片来源:诺贝尔奖官网)
下面,药明康德内容团队将与大家一道回顾这个革命性的AI解决方案的诞生和成长史。
AlphaFold的故事
首先,让我们来看两位共同主导了AlphaFold开发的获奖者的故事。其中,Demis Hassabis博士是DeepMind的创始人兼首席执行官,同时也是AlphaFold项目的负责人。John Jumper博士则是AlphaFold项目的首席高级研究员。深度学习算法使蛋白结构预测领域实现飞跃我们都知道,蛋白质是维持我们生命所必需的庞大而复杂的物质。我们身体的几乎所有功能,例如收缩肌肉、感知光线或将食物转化成能量等,都需要一种或多种蛋白质来完成。而蛋白质具体能做什么就要取决于它独特的3D结构了。然而,纯粹基于其基因序列推测蛋白质的3D结构是一项非常具有挑战性的复杂任务。这是因为我们的DNA通常只包含蛋白质中氨基酸残基的序列信息,而这些氨基酸残基形成的长链将会折叠成错综复杂的3D结构,加上蛋白质越大,需要考虑的氨基酸之间的相互作用就越多,对其结构的建模过程就会更加复杂和困难。在过去的数十年中,科学家们已经能够利用冷冻电子显微镜、核磁共振或X射线晶体学等技术在实验室中确定蛋白质的形状,但这些方法都需要通过大量的试错才能获得最终的结果,这可能需要花上好几年时间以及大量的资金。幸运的是,得益于基因测序成本的快速降低,基因组学领域的数据变得丰富了起来。一些科学家们开始利用AI技术开发深度学习算法,在基因组学数据的基础上对蛋白质结构进行预测。在此基础上,AlphaFold诞生了。 图片来源:123RF2018年12月,DeepMind宣布推出全新的AlphaFold系统,能够预测并生成蛋白质的3D结构。在当年的国际蛋白质结构预测竞赛(CASP)上,初次登场的AlphaFold就成为了最大的黑马,以绝对的优势击败了上百位的参会选手,拔得头筹。在比赛中,AlphaFold成功预测了给定的43种蛋白质中的25种的最准确结构,而同一类别的第二名参赛队伍只预测出了43种中的3种。和以往依赖预先构想逻辑的传统人工智能方法不同的是,AlphaFold并未使用已经明确结构的蛋白质3D模型作为模板,而是通过将机器学习作为蛋白质结构预测网络的核心组成部分,让AlphaFold从数据中自行发现模式规律。DeepMind团队使用的方法都以深度神经网络为基础,从基因序列中预测蛋白质的两种物理性质:氨基酸对之间的距离及连接这些氨基酸的化学键之间的角度。首先,研究小组训练了一个深度神经网络,来预测蛋白质中每对氨基酸残基之间距离的分布情况。然后,研究人员将这些数值转化为评分,来对蛋白质结构的精确程度进行评估。同时,研究人员还另外训练了一个神经网络,利用这些距离数值来评估预测结构与真实结构的接近程度。不仅如此,DeepMind的研究人员还在这些评分函数的基础上,使用了两种全新的方式来优化蛋白质结构评分:他们使用了一个生成神经网络,不断生成新的蛋白质片段来反复替换一段旧的蛋白质结构,这样一来,蛋白质结构的评分就被不断提高了。另外,研究人员还使用了一种名为梯度下降的方式来让AlphaFold预测的结构变得高度精确。梯度下降是一种机器学习中常用的数学技术,用来实现渐进式的细微改进。研究人员将这项技术用于整个蛋白质链,而不是结构中组装前必须分开折叠的片段,降低了预测过程的复杂性。AlphaFold再次进化加速科学发现尽管AlphaFold的首战告捷,但DeepMind的研究人员并不满意:他们希望得到一种对于实验人员更加有用的工具,误差小于1埃米(原子的大小)。经过多轮的调试和集思广益,DeepMind的研究团队在原来的算法基础上成功构建出了AlphaFold2。在2020年的CASP上,DeepMind的AlphaFold2系统表现惊艳,在接受检验的近100个蛋白靶点中,AlphaFold2对三分之二的蛋白靶点给出的预测结构与实验手段获得的结构相差无几。有些情况下,已经无法区分两者之间的区别是由于AlphaFold2的预测出现错误,还是实验手段产生的假象。
图片来源:123RF2018年12月,DeepMind宣布推出全新的AlphaFold系统,能够预测并生成蛋白质的3D结构。在当年的国际蛋白质结构预测竞赛(CASP)上,初次登场的AlphaFold就成为了最大的黑马,以绝对的优势击败了上百位的参会选手,拔得头筹。在比赛中,AlphaFold成功预测了给定的43种蛋白质中的25种的最准确结构,而同一类别的第二名参赛队伍只预测出了43种中的3种。和以往依赖预先构想逻辑的传统人工智能方法不同的是,AlphaFold并未使用已经明确结构的蛋白质3D模型作为模板,而是通过将机器学习作为蛋白质结构预测网络的核心组成部分,让AlphaFold从数据中自行发现模式规律。DeepMind团队使用的方法都以深度神经网络为基础,从基因序列中预测蛋白质的两种物理性质:氨基酸对之间的距离及连接这些氨基酸的化学键之间的角度。首先,研究小组训练了一个深度神经网络,来预测蛋白质中每对氨基酸残基之间距离的分布情况。然后,研究人员将这些数值转化为评分,来对蛋白质结构的精确程度进行评估。同时,研究人员还另外训练了一个神经网络,利用这些距离数值来评估预测结构与真实结构的接近程度。不仅如此,DeepMind的研究人员还在这些评分函数的基础上,使用了两种全新的方式来优化蛋白质结构评分:他们使用了一个生成神经网络,不断生成新的蛋白质片段来反复替换一段旧的蛋白质结构,这样一来,蛋白质结构的评分就被不断提高了。另外,研究人员还使用了一种名为梯度下降的方式来让AlphaFold预测的结构变得高度精确。梯度下降是一种机器学习中常用的数学技术,用来实现渐进式的细微改进。研究人员将这项技术用于整个蛋白质链,而不是结构中组装前必须分开折叠的片段,降低了预测过程的复杂性。AlphaFold再次进化加速科学发现尽管AlphaFold的首战告捷,但DeepMind的研究人员并不满意:他们希望得到一种对于实验人员更加有用的工具,误差小于1埃米(原子的大小)。经过多轮的调试和集思广益,DeepMind的研究团队在原来的算法基础上成功构建出了AlphaFold2。在2020年的CASP上,DeepMind的AlphaFold2系统表现惊艳,在接受检验的近100个蛋白靶点中,AlphaFold2对三分之二的蛋白靶点给出的预测结构与实验手段获得的结构相差无几。有些情况下,已经无法区分两者之间的区别是由于AlphaFold2的预测出现错误,还是实验手段产生的假象。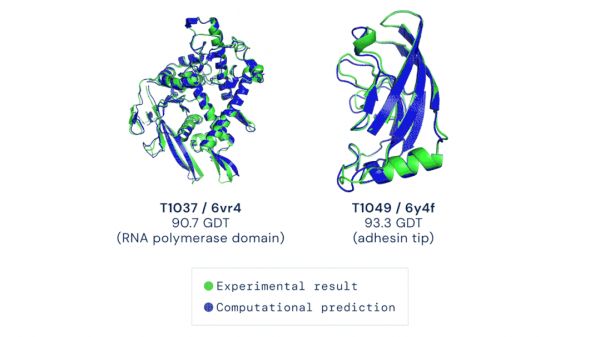 ▲AlphaFold2根据氨基酸序列预测的蛋白结构与实验手段解析的结果几乎完全重合(绿色,实验结果;蓝色,计算预测结果;图片来源:DeepMind Blog)2021年,Hassabis博士和Jumper博士与欧洲分子生物学实验室的欧洲生物信息学研究所(EMBL-EBI)合作,发布了AlphaFold预测的蛋白结构数据库(AlphaFold Protein Structure Database)。这个数据库涵盖了人类和20种常用模式生物的35万个蛋白质结构,并且对98.5%的人类蛋白质结构进行了准确预测——要知道在此之前,科学界解析的蛋白质结构只覆盖了人类蛋白序列17%的氨基酸。欧洲生物信息研究所主任Ewan Birney博士称该数据库为人类基因组图谱发布以来最重要的数据库之一。人工智能预测蛋白质结构领域的一系列突破,也被《科学》评选为2021年的年度科学突破。而更令人激动的是,他们开发的这一数据库将免费提供给全球的科研人员开放使用!许多科学家和生物医药公司的研究员兴奋地表示,这一系列突破将加速新药开发,并为基础科学带来全新革命。2022年,DeepMind与EMBL-EBI团队的合作又迎来了一项巨大的飞跃。AlphaFold对蛋白质结构的预测不再局限于人类与模式生物,而是拓展至涵盖了动植物、细菌等的100万个物种。不仅如此,其预测的蛋白质结构数量也提升了数百倍。AlphaFold2已对超过2亿种蛋白质进行了结构预测——几乎是科学界已知的所有蛋白质。同样的,这2亿种蛋白质的结构预测数据依然向公众免费开放,使研究人员能够像使用谷歌搜索信息一样搜索蛋白质的结构,为研究人员即时提供他们正在研究的任何蛋白质的预测模型,大大减少了他们曾经需要花在确定蛋白质结构上的时间。
▲AlphaFold2根据氨基酸序列预测的蛋白结构与实验手段解析的结果几乎完全重合(绿色,实验结果;蓝色,计算预测结果;图片来源:DeepMind Blog)2021年,Hassabis博士和Jumper博士与欧洲分子生物学实验室的欧洲生物信息学研究所(EMBL-EBI)合作,发布了AlphaFold预测的蛋白结构数据库(AlphaFold Protein Structure Database)。这个数据库涵盖了人类和20种常用模式生物的35万个蛋白质结构,并且对98.5%的人类蛋白质结构进行了准确预测——要知道在此之前,科学界解析的蛋白质结构只覆盖了人类蛋白序列17%的氨基酸。欧洲生物信息研究所主任Ewan Birney博士称该数据库为人类基因组图谱发布以来最重要的数据库之一。人工智能预测蛋白质结构领域的一系列突破,也被《科学》评选为2021年的年度科学突破。而更令人激动的是,他们开发的这一数据库将免费提供给全球的科研人员开放使用!许多科学家和生物医药公司的研究员兴奋地表示,这一系列突破将加速新药开发,并为基础科学带来全新革命。2022年,DeepMind与EMBL-EBI团队的合作又迎来了一项巨大的飞跃。AlphaFold对蛋白质结构的预测不再局限于人类与模式生物,而是拓展至涵盖了动植物、细菌等的100万个物种。不仅如此,其预测的蛋白质结构数量也提升了数百倍。AlphaFold2已对超过2亿种蛋白质进行了结构预测——几乎是科学界已知的所有蛋白质。同样的,这2亿种蛋白质的结构预测数据依然向公众免费开放,使研究人员能够像使用谷歌搜索信息一样搜索蛋白质的结构,为研究人员即时提供他们正在研究的任何蛋白质的预测模型,大大减少了他们曾经需要花在确定蛋白质结构上的时间。 图片来源:123RF目前,这些数据已经在疟疾疫苗开发、解决抗生素耐药性问题与塑料污染等场景中得到应用,并能够帮助研发人员加速新药研发。除此以外,该模型还具有加速生物学各个研究领域的潜力,其应用前景正等着更多才华横溢的科学家们来尽情开发。
图片来源:123RF目前,这些数据已经在疟疾疫苗开发、解决抗生素耐药性问题与塑料污染等场景中得到应用,并能够帮助研发人员加速新药研发。除此以外,该模型还具有加速生物学各个研究领域的潜力,其应用前景正等着更多才华横溢的科学家们来尽情开发。David Baker与Rosetta的故事
提到蛋白质从头设计,华盛顿大学蛋白质设计研究所所长David Baker博士的大名可谓是无人不知。不过,可能很少有人知道,在走上生物化学研究的道路前,Baker的专业是研究哲学。1983年,一堂关于蛋白质折叠问题的生物学课程彻底改变了他的人生轨迹。自那以后,这个众多科学家前赴后继尝试破解的生物学难题便成了他毕生的研究课题。小小的蛋白质里藏着天大的疑问
在生物体内,蛋白质让很多科学家们着迷。这种分子的尺寸只有纳米大小,复杂程度却可以超过任何一台人造的机器,大自然的精妙由此也可见一斑。1983年,在哈佛大学学习哲学的Baker在一堂生物学课程上了解到蛋白质折叠问题。此前的科学家们通过试验发现,这些复杂的蛋白质只由20种简单的氨基酸经过排列组合拼接而成,而一条氨基酸序列就已包含了它能形成蛋白质的所有结构和活性信息。就像有设计图纸一样,一条氨基酸序列可以自发折叠成唯一的三维结构,然后在细胞内发挥特定的功能——有的可以结合DNA,控制基因的开关;有的可以识别病原体,启动免疫反应。在这些现象背后,一个巨大的问题随之浮现:一条氨基酸序列从理论上来说可以有无数种折叠方式,那为什么它能够自发折叠成唯一的三维结构呢? 图片来源:123RF自那堂课后,Baker对这个数十年来困扰了无数科学家的难题产生了极大的兴趣,甚至不惜转换专业在生物学领域从头开始学习。而当他和导师提起他想要对这个难题发起挑战时,他的导师劝他不要头脑发热,因为“没人知道这是怎么回事”。听从了导师的建议,Baker将这一念头短暂封存,并在未来的诺奖得主Randy Schekman教授课题组获得了博士学位,主攻细胞生物学。博士后期间,Baker接触到使用计算机科学来进行结构生物学研究的方法。在这个过程中,他发现使用计算机解析晶体结构并不是他擅长的,但他却萌生出了另一个想法,或许计算机可以帮他实现那个他始终放不下的梦想——解开蛋白质折叠之谜。
图片来源:123RF自那堂课后,Baker对这个数十年来困扰了无数科学家的难题产生了极大的兴趣,甚至不惜转换专业在生物学领域从头开始学习。而当他和导师提起他想要对这个难题发起挑战时,他的导师劝他不要头脑发热,因为“没人知道这是怎么回事”。听从了导师的建议,Baker将这一念头短暂封存,并在未来的诺奖得主Randy Schekman教授课题组获得了博士学位,主攻细胞生物学。博士后期间,Baker接触到使用计算机科学来进行结构生物学研究的方法。在这个过程中,他发现使用计算机解析晶体结构并不是他擅长的,但他却萌生出了另一个想法,或许计算机可以帮他实现那个他始终放不下的梦想——解开蛋白质折叠之谜。向梦想靠近,开发出蛋白质结构预测程序
1993年,Baker成功获得了华盛顿大学生物化学系助理教授的职位,开始独立工作。在他招收第二个学生后,他建议学生借助计算机的力量做蛋白质结构预测相关的课题。1996年,他与研究生们开始编写一个叫做Rosetta的程序,这个程序有潜力根据一段氨基酸序列解出蛋白质的结构。在自然界中,为了保持稳定,蛋白质总是折叠成具有“最低自由能”的形状。这就好像水会从高处往低处流,然后停留在那里一样。不过利用计算机预测蛋白质结构也并没有想象中那么简单。由于每个氨基酸至少有三种不同的构象,那么一个仅含有100个氨基酸的蛋白质,其可能的结构就高达3的100次方种,这对计算机来说都是个难以处理的运算量。不过,Rosetta的程序设计用了一种十分巧妙的方法,它不是通过穷举法从这些天文数字般的可能结构中挨个寻找自由能最低的形状,而是先分析蛋白质的生物物理特性,模拟出一个大致的形状,然后进行微调,只留下自由能更低的结果。这样一来,研究人员们可以更快预测出蛋白质的结构。好消息是,Rosetta的表现十分惊艳。自1994年起,和Baker一样想要解开蛋白折叠之谜的生物学家们会定期聚在一起,检验各自的成果:就像考试一般,他们会拿到一个蛋白质的序列,然后预测出它的结构。随后,这些预测结构会和已通过实验方法得到解析但尚未公开的真实结构进行比对,看哪一个结构更为接近。在这个被誉为蛋白质结构领域“奥林匹克”的活动中,Rosetta程序总是最有力的竞争者,并且具有统治性的优势。开发Rosetta的意外收获
在Rosetta诞生的过程中,Baker还有许多意料之外的收获。尽管Rosetta的设计经过优化,但预测蛋白质折叠所需要的运算量依然巨大。最开始,Baker只能通过不停购置新的电脑设备来扩大计算力,后来,新买的电脑把实验室的空间占满了却依然无法满足他们的需求。迫于这样的压力,Baker和他的学生们想出了一个绝妙的解决方案——借助互联网,邀请世界各地的人们用他们计算机的闲置算力来帮助进行计算。2005年,Baker团队启动了一个名为Rosetta@home的项目,基于他们开发的Rosetta软件包,利用分布式计算的力量来解析蛋白质结构。令人感到意外的是,这些“网友”们还给Baker发去了反馈意见,表示计算机折叠没有他们手动折叠来得更好。更巧的是,当他与一名计算机科学家聊起这些话题时,俩人灵感迸发,决定从Rosetta@home出发开发一款游戏,让全世界对蛋白折叠感兴趣的人能够发挥他们的才华,参与到蛋白质折叠的解谜游戏中。这款名为Foldit的游戏由于能帮助学生更好地了解蛋白质的三维性质以及蛋白质结构和功能间的关系,已被一些大学引入课堂。更令人吃惊的是,一些该游戏的高级玩家还曾通过这款游戏破解了一种逆转录病毒的蛋白结构,并将成果发表在了《自然》杂志子刊上。 ▲Foldit玩家确定M-PMV逆转录病毒蛋白酶结构的过程(图片来源:参考资料[19])除此以外,与Foldit同时期诞生的还有一个名为Rosetta Commons的学术团体。这个团体的成员包括许多高校和研究机构的人员,其中很多都在Baker的实验室工作过。除了日常的交流合作,他们会定期举办会议分享最新成果、讨论如何进一步优化Rosetta,并开设训练营培训那些对Rosetta感兴趣但不知道如何使用的人。
▲Foldit玩家确定M-PMV逆转录病毒蛋白酶结构的过程(图片来源:参考资料[19])除此以外,与Foldit同时期诞生的还有一个名为Rosetta Commons的学术团体。这个团体的成员包括许多高校和研究机构的人员,其中很多都在Baker的实验室工作过。除了日常的交流合作,他们会定期举办会议分享最新成果、讨论如何进一步优化Rosetta,并开设训练营培训那些对Rosetta感兴趣但不知道如何使用的人。从预测到模拟,破解上帝之手的奥秘
虽然Baker最初的研究方向是预测蛋白质的结构,但在这个方向上取得突破之前,他已着手向另一个截然相反且更具挑战性的领域——“蛋白质的从头设计”发起了冲击。相比于预测蛋白质的结构,从头设计出一个蛋白质需要向弄清蛋白质折叠的原理再迈进一步。这要求科学家们能根据一个具有特定形状的蛋白,倒推出其DNA序列。从某种意义上讲,从头设计蛋白,要比预测蛋白结构难上几个数量级。假设要设计一个由100个氨基酸组成的蛋白质,每一种氨基酸又有20种截然不同的可能,使将得可能的氨基酸序列总数高达20的100次方。这个数字究竟有多大?它比整个宇宙中原子的总数还要多!由于Baker在Rosetta的开发中已经取得过一定的经验,这次再开发从头设计蛋白质的方法就有了良好的基础。从DNA序列到蛋白质结构,Rosetta能找到能量最低的形状。那么反过来,Rosetta也能用来推导为了构成这一形状所需的蛋白组件。在此基础上,研究人员们还学会了如何像拆解乐高玩具一样,将一个蛋白质拆成螺旋或者桶装的小块,分块击破。 图片来源:123RF2003年,Baker的团队设计出了第一个原本并不存在于自然界中的蛋白质,它被命名为Top7。这当然是一个重要突破,但却没有开辟一个崭新的时代。Baker实验室的成员开玩笑说Top7只是一块从热力学角度上看很稳定的“石头”。因为他们从头设计出的这个蛋白质虽然折叠成了研究人员们想要它折叠的模样,但不具有任何功能。7年后,Baker的一名博士后研究员做出了改进。他将抗体的一部分连接到了人造蛋白上,使人造蛋白首度具有了功能:新合成的蛋白能识别流感病毒,有望成为一种新的药物,但这多少有些“作弊”的意思,毕竟最重要的那部分来自天然的抗体。接下来的几年时间,Baker的团队对Rosetta进行了更多的优化。如今,Baker的实验室,以及他的合作伙伴们已能设计出多种不同的蛋白,有朝一日,人类完全获得“上帝之手”的能力将不再是梦想。不过到目前,从头设计蛋白依然是一个不断试错的工作,需要大量的资源投入。以设计结合蛋白为例,从流程上看,科学家会首先用Rosetta模拟出所感兴趣蛋白表面上的一个“口袋”,然后再设计出大量不同的螺旋结构,形成稳定骨架。这些骨架上含有一些特定的氨基酸,有可能会与“口袋”进行完美的契合。这个工作就像是在一把钥匙上不断打磨,最终使其完美地对应一把锁。随后,研究人员们会根据设计合成所要的DNA序列,将其引入细菌细胞,期望它们能够产生所需要的蛋白。获取这些蛋白后,他们还会做两个测试:评估这些蛋白是否能如预期般折叠,以及折叠后的蛋白是否能如预期般结合特定蛋白。通常来讲,人工设计的蛋白极少能同时满足这两个条件。而那些脱颖而出的蛋白,则会成为新一轮设计与筛选的起点,直至获得最佳的构象。
图片来源:123RF2003年,Baker的团队设计出了第一个原本并不存在于自然界中的蛋白质,它被命名为Top7。这当然是一个重要突破,但却没有开辟一个崭新的时代。Baker实验室的成员开玩笑说Top7只是一块从热力学角度上看很稳定的“石头”。因为他们从头设计出的这个蛋白质虽然折叠成了研究人员们想要它折叠的模样,但不具有任何功能。7年后,Baker的一名博士后研究员做出了改进。他将抗体的一部分连接到了人造蛋白上,使人造蛋白首度具有了功能:新合成的蛋白能识别流感病毒,有望成为一种新的药物,但这多少有些“作弊”的意思,毕竟最重要的那部分来自天然的抗体。接下来的几年时间,Baker的团队对Rosetta进行了更多的优化。如今,Baker的实验室,以及他的合作伙伴们已能设计出多种不同的蛋白,有朝一日,人类完全获得“上帝之手”的能力将不再是梦想。不过到目前,从头设计蛋白依然是一个不断试错的工作,需要大量的资源投入。以设计结合蛋白为例,从流程上看,科学家会首先用Rosetta模拟出所感兴趣蛋白表面上的一个“口袋”,然后再设计出大量不同的螺旋结构,形成稳定骨架。这些骨架上含有一些特定的氨基酸,有可能会与“口袋”进行完美的契合。这个工作就像是在一把钥匙上不断打磨,最终使其完美地对应一把锁。随后,研究人员们会根据设计合成所要的DNA序列,将其引入细菌细胞,期望它们能够产生所需要的蛋白。获取这些蛋白后,他们还会做两个测试:评估这些蛋白是否能如预期般折叠,以及折叠后的蛋白是否能如预期般结合特定蛋白。通常来讲,人工设计的蛋白极少能同时满足这两个条件。而那些脱颖而出的蛋白,则会成为新一轮设计与筛选的起点,直至获得最佳的构象。遇劲敌,取长补短开启新一轮进化之路
在2018年以前,Baker及其团队开发的Rosetta在蛋白质结构预测领域完全没有对手。而那一年,AlphaFold的出现令Baker嗅到了危机。尽管18年的蛋白质结构预测竞赛依然是Rosetta拔得头筹,但首次亮相就获得了第二名的AlphaFold令Baker见识到了机器学习的过人之处。于是,他要求团队紧跟时代的风向,加紧研究机器学习。Baker的预感没有错,在2020年的竞赛中,第二代AlphaFold击败了Rosetta,一举成名。不过,Baker率领着团队很快就追赶了上来。2021年7月15日,当DeepMind公司在《自然》杂志上发表论文,公开了“AlphaFold2”的源代码,并且详细描述了它的设计框架和训练方法时,Baker的团队也于《科学》杂志上介绍了其开发的RoseTTAFold算法。RoseTTAFold的神经网络能够同时考虑蛋白序列的模式、蛋白中不同氨基酸之间的相互作用,以及蛋白质可能出现的3D结构。在这个系统中,一维、二维和三维的信息能够相互交流,让神经网络综合所有信息,决定蛋白质的化学组成部分和它折叠产生的结构之间的关系。 ▲RoseTTAFold系统结构简介(图片来源:参考资料[12])研究人员表示,RoseTTAFold系统在解析蛋白质3D结构方面的表现与AlphaFold2的水平几乎相当,在有些蛋白上甚至优于AlphaFold2。利用来自AlphaFold的公开信息,也得益于多年来对于机器学习的积累,这个算法的开发只用了区区几个月的时间。
▲RoseTTAFold系统结构简介(图片来源:参考资料[12])研究人员表示,RoseTTAFold系统在解析蛋白质3D结构方面的表现与AlphaFold2的水平几乎相当,在有些蛋白上甚至优于AlphaFold2。利用来自AlphaFold的公开信息,也得益于多年来对于机器学习的积累,这个算法的开发只用了区区几个月的时间。下一站,剑指新药开发
作为蛋白质从头设计的先驱者,Baker希望通过“蛋白质设计革命”开启一个全新的时代,我们将学会使用一种前所未有的方式来操控生物分子,例如从头设计出全新的药物、疫苗、疾病疗法等,拓展新药研发的边界。2022年8月,Baker及其团队在《细胞》杂志上发表论文,他们已利用AI技术平台精准地从头设计出能够穿过细胞膜的大环多肽分子,开辟了设计全新口服药物的新途径。同时,Baker团队成员联合创建的初创公司Vilya也正式亮相,并从著名风投机构ARCH Venture Partners获得5000万美元A轮融资。利用这一技术,跳过高通量筛选、直接合成候选药物的策略不再遥不可及!今年以来,Baker及其团队已在《自然》和《科学》杂志上发表了数篇重磅论文,其开发的全新的蛋白质从头设策略法可靶向不可成药靶点,并能实现按需设计生物分子,为蛋白设计提供了更广阔的可能性。 华盛顿大学蛋白设计研究所首席战略及运营官Lance Stewart博士是David Baker教授长期的合伙伙伴。在2023年药明康德全球论坛上,他指出:“现在的新技术让我们有能力去挑战任何类型的靶点,这是当下生物医药产业的幸运。”在获得此次诺贝尔奖之前,Demis Hassabis博士与John Jumper博士还在今年获得了多个科学大奖,包括盖尔德纳奖(Gairdner)和拉斯克奖。2020年,Baker博士也获得了素有“科学界的奥斯卡”之称的科学突破奖——生命科学科学突破奖。此次共同摘得诺贝尔奖的桂冠是对他们通过放飞想象力和才华,解决了让科学家困惑了半个世纪的蛋白质结构预测难题的再次肯定。让我们再次祝贺这三位杰出的科学家,并向他们突出的贡献致以崇高敬意!参考资料(可上下滑动查看)
华盛顿大学蛋白设计研究所首席战略及运营官Lance Stewart博士是David Baker教授长期的合伙伙伴。在2023年药明康德全球论坛上,他指出:“现在的新技术让我们有能力去挑战任何类型的靶点,这是当下生物医药产业的幸运。”在获得此次诺贝尔奖之前,Demis Hassabis博士与John Jumper博士还在今年获得了多个科学大奖,包括盖尔德纳奖(Gairdner)和拉斯克奖。2020年,Baker博士也获得了素有“科学界的奥斯卡”之称的科学突破奖——生命科学科学突破奖。此次共同摘得诺贝尔奖的桂冠是对他们通过放飞想象力和才华,解决了让科学家困惑了半个世纪的蛋白质结构预测难题的再次肯定。让我们再次祝贺这三位杰出的科学家,并向他们突出的贡献致以崇高敬意!参考资料(可上下滑动查看)[12] Baek, Minkyung et al. (2021) Accurate prediction of protein structures and interactions using a three-track neural network. Science. vol. 373,6557: 871-876. DOI:10.1126/science.abj8754
[15] Pan, Xingjie, and Tanja Kortemme. (2021) Recent advances in de novo protein design: Principles, methods, and applications. The Journal of biological chemistry 296: 100558. DOI:10.1016/j.jbc.2021.100558
[16] Mirdita, Milot et al. (2022) ColabFold: making protein folding accessible to all. Nature methods 19,6: 679-682. DOI:10.1038/s41592-022-01488-1
[18] Silva, Daniel-Adriano et al. (2019) De novo design of potent and selective mimics of IL-2 and IL-15. Nature 565,7738: 186-191. DOI:10.1038/s41586-018-0830-7
[19] Khatib F, DiMaio F; Foldit Contenders Group; Foldit Void Crushers Group; Cooper S, Kazmierczyk M, Gilski M, Krzywda S, Zabranska H, Pichova I, Thompson J, Popović Z, Jaskolski M, Baker D. Crystal structure of a monomeric retroviral protease solved by protein folding game players. Nat Struct Mol Biol. 2011 Sep 18;18(10):1175-7. doi: 10.1038/nsmb.2119. Erratum in: Nat Struct Mol Biol. 2012 Mar;19(3):364. PMID: 21926992; PMCID: PMC3705907.
免责声明:药明康德内容团队专注介绍全球生物医药健康研究进展。本文仅作信息交流之目的,文中观点不代表药明康德立场,亦不代表药明康德支持或反对文中观点。本文也不是治疗方案推荐。如需获得治疗方案指导,请前往正规医院就诊。
分享,点赞,在看,聚焦全球生物医药健康创新
相关知识
2022年诺贝尔奖,将花落谁家?获奖预测!@MedSci
分子系统生物学研究中心举办第二十六期未来科学沙龙中国科学院遗传与发育生物学研究所
两名科学家因“基因剪刀”获得诺贝尔化学奖
关怀人文、赋能科研 科大讯飞展示AI如何改变生活 — 新京报
2024年诺贝尔奖即将揭晓!花落谁家?这几个“风向标”或许暗藏线索
两位科学家因发现miRNA及其调控作用获奖
AI中医香方仪 | OPENAIGC开发者大赛企业组AI创作力奖
上海首发四款垂直大模型,聚焦AI与基础科学研究的融合发展
讯飞星火开发者生态迅猛发展,科大讯飞携生态伙伴引领大模型规模化落地
张锋赢得专利、憾失诺奖,细数CRISPR基因编辑三巨头之间的爱恨情仇
网址: 诺贝尔化学奖也花落AI领域!AlphaFold开发者等人获奖 https://m.huajiangbk.com/newsview627766.html
| 上一篇: 【科学网】它凭啥占诺贝尔物理学奖 |
下一篇: 诺贝尔经济学奖授予阿西莫格鲁等3 |