成功案例-通过全基因组重测序揭示莲的进化模式
莲属(Nelumbo Adans)是莲科,山龙眼目,双子叶植物。虽然莲属是莲科唯一的属,它也只包括两个种:Nelumbo nucifera Gaertn (Asian lotus or sacred lotus)和Nelumbo lutea Pers(American lotus)。这两个种类由太平洋分开,但它们保持相同的染色体数目(2n = 16)。比较转录组研究表明在叶和根茎发育过程中这两种生态型之间存在较多的转录差异。 由于热带莲花的这些特征,它经常被用于通过杂交改善温带莲花的性状。 因此,了解莲的基因组多样性和亲缘关系对重要种质资源的育种以及原位和非原位保存有重要意义。
英文题目:Whole genome re-sequencing reveals evolutionary patterns of sacred lotus ( Nelumbo nucifera )
发表杂志:Journal of Integrative Plant Biology
PMID:29052958
莲(sacred lotus)基因组草图的发布对莲的研究产生了深远的影响。 通过基因组重测序分析热带莲(清迈野莲)和温带莲(中湖野生莲)之间的关系,鉴定了大量的基因组变异,包括SNPs,InDels和SVs。 然而,莲花基因组在不同地理来源和不同育种目的的情况下如何进化仍不清楚。
为了探索与根茎生长模式相关的基因组多样性,微进化,特别是生态型分化的基因组标记,中国科学院武汉植物园石涛老师课题组研究人员对19种莲进行基因组重测序和分析,包括根茎莲,种子莲,花莲, 温带莲,热带莲和美国莲。通过对重测序数据的全面分析,为不同种质资源莲的遗传进化提供可靠和详细的认知,并为根茎扩大的关键突变提供线索,用于种质资源保存和分子育种研究中。
材料和方法
材料:18个亚洲莲(Nelumbo nucifera Gaertn),(包括2个根茎莲,2个荷花莲,2个种子莲,7个野生莲,5个泰国莲)和1个美国野生莲(Nelumbo lutea Pers),其中泰国莲属于热带莲,其余的亚洲莲均属于温带莲花,收集自中国科学院武汉植物园(中国武汉)。收获嫩叶并通过CTAB方法进行基因组DNA提取,建库测序(表1)
测序方法:基因组重测序
测序平台:Illumina HiSeq 2500平台350bp
分析平台与内容
分析平台:BMKCloud(www.biomarker.net)
公共数据获取:从NCBI下载泰国莲花“清迈野生莲”重测序数据,登录号为SRP061673。
分析内容:
1.通过BWA与莲花参考基因组'China Antique'进行比对,突变检测。
2.突变过滤,验证和注释
3.系统发育,群体分层和PCA分析
4.全基因组比较基因突变率
5.鉴定与生态分化相关的候选基因
结果
1、基因组变异
19个样本平均测序深度为4×,≥1×覆盖度超过99%。所有变异信息如表1所示。通过基因组变异与之前公布的相应转录组的变异进行比较来验证基因组重测序的准确性,由于InDels可以通过转录本的可变剪接或mRNA降解导致,所以变异的最终一致率由SNP的一致率表示。结果显示‘Zhouou’基因组重测序与自身转录组数据中SNP的一致率为88.79%。这些基因组与转录组数据的比较证实了变异的有效性,且系统发育信号存在于编码序列区域中。
表1.所有莲样本的变异信息
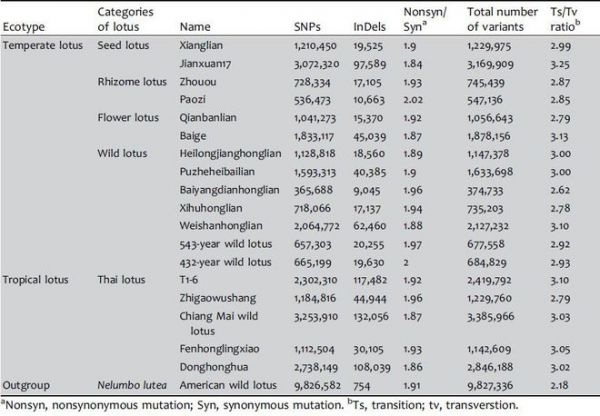
一般来说,不同的莲个体检测到的变异数不同。各样本比较后发现,美洲野生莲,显示出最高的变异数(9,826,582)和最低的转换 - 颠换率(Ts / Tv = 2.18),这可能是由于长期大陆隔离没有基因交换导致的。除美国野生莲外, “Jianxuan-17 ” 和“Chiang Mai wild lotus”表现出更多的变异。两种根茎莲和四种野生莲表现出较少的变异(表1)。且个体间非同义和同义替换的比率(Nonsyn / Syn)也不同。种子莲的“Jianxuan17” 具有最低比率(Nonsyn / Syn = 1.84),而一种根茎莲“Paozi” 比率最高(Nonsyn / Syn = 2.02)(表1)。此外,研究人员还发现莲比桃子(1.65),大豆(1.61)和大米(1.29)具有更高的Nonsyn / Syn比率,表明莲中非同义突变的比例相对较高。
2、种群结构
为了阐明各样本之间的遗传关系,基于这19个个体的非缺失SNP位点进行种群分层分析和PCA(主成分分析)(图1A-C)。基于两种SNP位点(非缺失SNP位点和4dSNP位点)构建两种系统发育树。通过系统发育分析发现,野生莲与其他的莲有复杂亲缘关系(图1A)。4种野生莲,“432-year wild lotus”,“543-year wild lotus”,“Baiyangdianhonglian”和“Xihuhonglian ”形成了一个独立的进化支,而根茎莲,“Paozi”和“Zhouou”被聚为一组,且与4种野生莲密切相关,这表明根茎莲和大部分野生莲的亲缘关系很近(图1A)。然而,其他3种野生莲“ Weishanhuhonglian ”“ Puzheheibailian”和“Heilongjianghonglian”中的bootstrap支持数非常低,不足以确定与其他莲的亲缘关系(图1A)。野生莲的复杂关系可能是自然栖息地丧失和栽培莲遗传渗入的结果,使得自然保护区的一些莲变成半野生。与此同时,泰国的3种莲,T1-6,“ Zhigaowushang ”和“ Fenhonglingxiao ”聚集在一起,bootstrap支持数为100。但是,另外2种泰国莲“Donghonghua”和“Chiang Mai wild lotus”分别与种子莲“Jianxuan17”和花莲“Qianbanlian”分成一组。由非缺失SNP位点和4d SNP位点构建的系统发育树有部分重合。
此外,通过STRUC-TURE进行种群分层分析,并且对19个样本进行PCA分析(图1B,C)。 结果显示,美国野生莲和亚洲莲之间存在很强的遗传分化。 因此,研究者将美国野生莲与其他莲建立了系统发育树。结果证实,根茎莲与野生莲亲缘关系近,而花莲和种子莲表现出了基因混合现象。

图1.19个样本的系统发育树,种群分层,PCA分析结果
3、多样化过程中的基因和基因家族进化
蛋白编码序列的突变可能会影响基因功能和生物学过程,因此研究人员通过测量功能基因的突变率(MR)来比较功能基因的进化模式。亚洲莲的蛋白编码基因的变异率通过变异密度统计。根据MR的四分位数范围将莲的基因进一步分为四组:MR非常低(6675个,MR≤1.5724),MR低(6668个,1.5724<MR≤3.4388),MR高(6,772,3.4388<MR≤6.3132),MR非常高(6,670个,MR>6.3132)。对每组进行GO富集分析表明,以不同比率进化的基因可能在其功能类别中有偏差(图2)。那些MR非常低的基因,分别被富集基因表达,RNA代谢过程调控等GO terms上(图2)。那些低MR的基因被富集到碳水化合物生物合成和代谢过程,酒精代谢过程等GO terms中(图2)。

图2.MR值四个等级(非常低、低、高、非常高)的基因GO富集图( P -value < 0.01)
在蛋白编码区,无义突变,缺失和成熟密码子终止等突变可能损害基因的功能。因此,研究人员比较了12个主要基因家族(包括植物免疫相关基因家族,如NB-ARC,蛋白激酶,富含亮氨酸的重复N末端和其他基因家族)中预测的功能缺失多态性(图3)。一般而言,这些基因家族中功能丧失突变率有所不同(22.4%-44.9%)(图3)。NB-ARC家族成员中功能丧失突变的比例最高(44.9%),其次是细胞色素P450(40.2%),NB-ARC的功能丧失率最高可能与基因流通率高有关,这也验证了NB-ARC在植物 - 病原体分支比赛中快速进化中的重要性。相反,基因转录相关家族,如环形手指(26.9%),DNA双螺旋结合(27.0%),Myb-Like(25.5%)和AP2结构域(23.8%)功能丧失突变有相对较低的比率(图3)。因此,研究者得出结论,由于功能上的差异,莲基因家族之间的进化动态特别是功能缺失突变的速率不同。
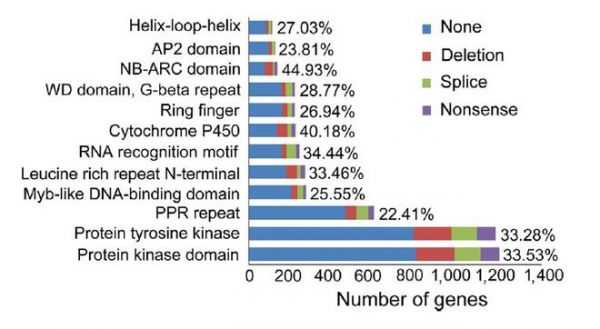
图3. 莲的12个主要基因家族中影响较大的突变
4、与生态分化相关的目的基因
为了鉴定温带和热带莲的分化基因区域和基因,根据记录的来源和根茎表型(图1,表1),在排除较强混合性个体后,在18个亚洲莲中选取14个样本分为两组。根据全基因组SNP数据计算两组之间的FST值以选择与生态分化相关的候选基因区域(图4)。FST的阈值设置为0.315001,总共筛选了5,341个候选基因(占莲总基因的20.01%)。为了进一步研究这些基因在进化方面是如何选择的,使用Tajima’s D对选取的14个亚洲莲进行计算(windows 10kb,step 5kb)。一般来说,Tajima's D与最小差异基因组区域内的基因相比,Tajima's D更高,并偏向候选基因的正值。这表明候选基因可能在平衡选择或频率依赖性选择方面略强。
此外,研究者对他们之前发布的三个根茎莲在温带和热带环境中初始发育阶段和快速发育阶段的表达模式进行了探索,研究基因序列分化是否与增加的差异表达和表型相关。在这些候选基因中,根茎发育相关阶段,温带和热带莲之间差异表达(FC不低于2)的基因有3,669个(68.70%),而在芽发育阶段有2,122(39.73%)个差异表达基因。发育阶段,共有4,063(76.07%)个候选基因在温带和热带莲之间有显著的差异表达,其比例显着高于注释基因组中差异表达基因的比例(74.57%)。这表明候选基因倾向于在两种生态型之间差异表达。
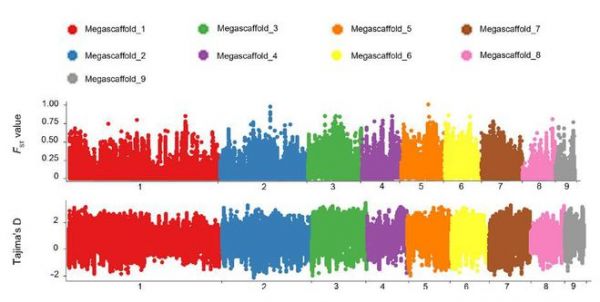
图4. 候选基因组区域用于温带和热带莲分化的鉴定
5、五个组之间遗传多样性和分化
研究人员将亚洲莲的18个个体根据其来源和园艺分类划分为五个组(野生,泰国,根茎,花和种子莲),研究人员总结了这些变异,并检测了各组之间的遗传多样性和分化,这对莲的育种有重要意义。结果显示:尽管野生莲每个个体之间的变异较少,但野生莲的总变异数量(6,424,745)远远高于其他组,反映了野生莲样品的地理位置和遗传多样性较丰富。在栽培的莲花中,根茎莲花的总变异最少(1,154,765),其次是花莲(3,020,048)和种子莲(4,094,993)。在维恩图中进一步描述了莲不同亚群之间的共有和特有的变异。泰国莲和野生莲的独有SNP和InDel比例较高,而根茎莲,花莲和种子莲的独有变异比例比较低。这些证实了,与栽培莲相比,野生莲和泰国莲的遗传多样性可能更高,泰国莲与野生莲之间的共有变异数最高,而根茎莲和种子莲或花莲是最低的。
为了描述这五个亚种的遗传多样性,研究者计算了基于SNP变异的核苷酸多样性(π)和其他遗传参数。所有栽培种中,根茎莲花的核苷酸多样性最低,而花莲和种子莲的核苷酸多样性较高。泰国莲表现出最高的核苷酸多样性,其次是野生莲花。栽培莲的遗传多样性降低表明它们在栽培期及人工选择时可能经历了种群瓶颈效应。根茎莲和野生莲具有较高的非同义/同义突变比率,但具有较低的转换率。根据前期的研究,说明这两个亚群可能在频繁无性繁殖中积累了更多的轻微有害突变。
为了评估这五个亚群之间的种群分化,研究者计算了全基因组水平上成对FST值。结果表明野生莲和根茎莲是所有配对比较中分化程度最低的亚群,这进一步支持了上述系统发育,群体分层和PCA分析结果。 有趣的是,在三个栽培亚组中,根茎莲和种子莲的种群分化最高,根茎和花莲呈中等分化,而花莲和种子莲呈现出最低分化。
结论
莲是园艺和生态系统中重要的水生植物。作为探索不同种质之间基因组变异和进化的基础,研究人员对19个样本进行了重测序分析,通过在全基因组中广泛分布的非缺失SNP位点进行遗传多样性和多态性分析,结果显示野生莲和泰国莲比栽培莲具有更高的遗传多样性。根茎莲具有最低的遗传多样性并且与野生莲的关系更密切,而种子莲和花莲的基因组存在混合。在能量代谢过程和植物免疫中的基因迅速进化,反映了一种局部适应现象。研究人员表明,温带和热带莲相关候选基因有显著差异,它们往往表现出高度不同的表达模式。总之,这项研究全面而可信地解释了莲的生态型分化的遗传多样性,亲缘关系,遗传进化和遗传标记的重要模式。
创新点
在以往的研究中,莲种质资源间的遗传多样性和亲缘关系研究主要通过ISSR,RAPD,AFLP和SSR等分子标记的方法进行,并且莲基因组在不同地理来源和不同育种目的的情况下如何进化仍不清楚。该研究采用高通量重测序的技术手段,对19种莲进行基因组重测序和分析,充分利用高通量SNP标记具有密度高,基因区覆盖率高,基因组分布广等优点,为不同种质资源莲的遗传进化提供可靠和详细的认知,并为根茎扩大的关键突变提供线索,对种质资源保存和分子育种研究做出重大贡献。
参考文献:
Huang L, Yang M, Li L, et al. Whole genome re-sequencing reveals evolutionary patterns of sacred lotus (Nelumbo nucifera).[J]. Journal of Integrative Plant Biology, 2017.
相关知识
成功案例-通过全基因组重测序揭示莲的进化模式
园艺作物基因组测序研究进展
世界首个梅花全基因组重测序研究完成
园艺作物基因组测序的研究进展.doc
基因测序研究揭秘兰花进化
揭示花椒基因组背后适应性进化机制
“深圳拟兰基因组与兰花的进化”重大科研成果新闻发布会
【学术前沿】睡莲基因组揭示早期开花植物的进化奥秘
分析染色体级别的基因组装配揭示了六倍体栽培菊花的起源和进化
江苏里下河地区农科院与福建农林大学合作完成春兰全基因组测序并揭示兰花观赏性状的分子调控机制
网址: 成功案例-通过全基因组重测序揭示莲的进化模式 https://m.huajiangbk.com/newsview253453.html
| 上一篇: 植物花粉发育相关基因的研究进展 |
下一篇: 秦跟基课题组揭示植物绿色花瓣形成 |