The Evolution Path of Medicago in China based on the Chloroplast Genome Analysis
摘要: 全球苜蓿属植物约87种, 我国有15种, 大多为广布种, 可生活在不同的生境当中。该属植物由于荚果等形态变异复杂, 繁育系统多样, 其进化历史一直是本领域关注的热点问题之一。随着分子生态学的发展, 利用基因组学的方法解决生态学问题不断报道, 但目前我国苜蓿属物种的叶绿体基因组数据缺乏, 有关其演化路径的研究尚少有开展。为此, 选取了我国苜蓿属10个代表物种和胡卢巴属1种, 测序比较其叶绿体基因组结构特征, 基于叶绿体基因组和核ITS序列构建分析其系统发育关系, 并结合物种地理分布生境特征探究我国苜蓿属植物的演化路径。结果显示, 以上苜蓿属植物的叶绿体基因组大小为121—127 kb, 均为非典型的“四区”结构, 缺失反向重复区。在10个苜蓿属物种的叶绿体基因组中共检测到1, 273个SSR位点, 这些SSR位点可以作为潜在的分子标记用于我国苜蓿属植物资源的鉴定。供试苜蓿属物种的叶绿体基因组共发生6次基因倒位。天蓝苜蓿的2个倒位发生在基因atpB到ycf3、ndhC到trnLUAA之间的区域;青海苜蓿和花苜蓿的叶绿体基因组共享4个倒位, 分别位于基因psbM到psaA、ndhB到trnNGUU、ndhB到rpoA以及clpP到rpl20区间。天蓝苜蓿中还存在clpP、rpoC1和atpF基因内含子的丢失现象。基于叶绿体基因组及ITS序列构建的苜蓿属系统发育关系, 支持传统上将苜蓿属划分为紫苜蓿组、南苜蓿组、阔荚苜蓿组和天蓝苜蓿组的分类处理。其中, 直果胡卢巴、单花胡卢巴与毛荚苜蓿形成一支, 位于系统树的基部, 支持中国苜蓿属和胡卢巴属之间存在过渡类型“类苜蓿植物”的观点。分析我国苜蓿属植物的分化原因可能与这些物种的生境有关, 特别是年平均温度可能是导致苜蓿物种分化的决定性因素。
Abstract: Medicago L. is a genus comprising approximately 87 species in the world, and 15 species in China. Most of them are widely distributed and can live in different habitats. Due to complex morphological variation such as pods, and diverse breeding systems, the evolutionary history of this genus has always been one of the hotspot. With the development of molecular ecology, the use of genomics to solve ecological problems has been continuously reported. However, the current chloroplast genome data of the genus of Medicago in China is still lacking, and the analysis of its evolution path has seldom carried out. In this study, ten Medicago species and one Trigonella species in China were selected to sequence and compare the structural characteristics of the chloroplast genomes. The phylogenetic relationship based on the chlroplast genome and nuclear ITS sequence were analyzed, and the evolution path were explored based on their geographical distribution and habitat characteristics. The results showed that the chloroplast genomes of the above species were 121—127 kb in size, all of which displayed atypical quadripartite structure missing one copy of the inverted repeat region (IR). Totally, 1, 273 variable SSR loci were found in these Medcago species, which could be used as potential molecular markers to identify Medicago germplasms. There were 6 inversions happened in the chloroplast genome of the tested Medicago species. Two unique inversions were identified between genes atpB to ycf3, ndhC to trnLUAA in M. lupulina. And four inversions were shared by M. archiducis-nicolai and M. ruthenica between genes psbM to psaA, ndhB to trnNGUU, ndhB to rpoA, clpP to rpl20. Intron losses were also observed in clpP, rpoC1 and atpF genes in M. lupulina chlroplast plastome. The phylogenetic relationship based on chloroplast genome and ITS sequences supported the traditional classification to subdivide Chinese Medicago into section Medicago, Spirocarpos, Platycarpae and Lupularia. The basal position of the clade consisting of M. edgeworthii, M. orthoceras and M. monantha like the Trigonella supported the existence of ″Trigonelloid″ Medicago. The differentiation may be related to the habitats of these species, especially the annual average temperature may be the decisive factor leading to the differentiation of Medicago in China.
Key Words: Medicago Plastome Structure variation Phylogenomics Ecological evolution
苜蓿属(Medicago Linn.)隶属于豆科蝶形花亚科车轴草族, 属下物种经历数次划分调整, 物种数量从最初的46种不断增加至目前的87种, 我国有15种[1—3]。苜蓿属植物的分类困难与其起源、地理分布以及相似的易混淆物种有关, 例如, 区分苜蓿属、胡卢巴属(Trigonella Linn.)和草木犀属(Melilotus Linn.)的一些物种, 一直是令人棘手的难题[4—5]。1977年, Ivanov[6]提出苜蓿属植物的起源地在高加索地区, 之后有学者认为是地中海北部海岸[1]。目前发现的苜蓿属大多数物种分布于地中海沿线、阿拉伯半岛、伊拉克和巴尔干东部地区, 而部分成员如紫花苜蓿复合体中的少数种则因栽培逃逸扩展至中亚、北亚和东亚地区[2—3]。苜蓿属植物为一年生或多年生草本, 稀灌木, 在整个生态系统乃至农牧生产系统中均具有重要的功能, 广泛分布于山地、农田、草地、森林等不同生态系统类型, 其中, 紫花苜蓿因其高蛋白、全营养和耐消化等优良特性, 成为世界上最为重要的饲料作物之一[7];截形苜蓿因其为二倍体、自花授粉、种子量大和遗传操作方便等特性, 成为豆科植物研究的模式植物[8]。
据《中国植物志》记载, 我国现有苜蓿属植物15种, 隶属于木本苜蓿组、天蓝苜蓿组、紫苜蓿组、阔荚苜蓿组和南苜蓿组, 而《Flora of China》未做属下等级的划分[2]。邓宏超等[4]研究了国内苜蓿属和胡卢巴属的分类划分, 并基于形态学特征等将苜蓿属植物分为三类:一是开紫花而荚果螺旋盘卷, 二是开黄花而荚果镰刀状弯曲或稍直, 三是花杂色而荚果镰刀状弯曲至螺旋盘卷的过渡类型。但由于苜蓿属植物耐受性强, 分布广泛, 生活型多变, 导致学界难以将苜蓿属植物的进化历史与其地理、生境特征直接关联。如何通过科学方法探究苜蓿属植物的生态演化路径已经成为本学科关注的热点之一。
随着分子生态学的出现和发展, 利用基因组学和分子生物学的方法解决生态学问题不断报道, 但目前我国有关苜蓿属植物叶绿体基因组和系统发育方面的研究尚开展较少, 通过叶绿体基因组揭示该属植物的起源、演化方面的研究也少有报道。叶绿体是细胞内一类具有自主遗传功能的重要细胞器。自1986年叶绿体基因组首次被测定以来[9—10], 已有近6, 549个物种的叶绿体基因组被测序, 涵盖大多数重要的真核光合类群(National Center for Biotechnology Information, NCBI, 2021年6月8日登录)。叶绿体基因组数据的迅速增加, 表明本领域已经逐渐成为研究热点, 同时也为揭示叶绿体基因组在研究生态进化中的重要价值提供了依据。研究表明, 叶绿体基因组不仅在探究物种起源、演化以及物种间亲缘关系等方面具有重要贡献[11—12], 而且在作物遗传改良、生物制剂生产等方面也显示出了巨大潜力[13]。基于此, 为了阐明我国苜蓿属植物的分子生态学结构特征, 探究其在我国的生态进化路径, 本研究选取我国10个苜蓿属代表物种和葫芦巴属一个物种作为研究对象, 采用高通量测序平台测定其叶绿体全基因组和核基因ITS序列, 通过分析其叶绿体基因组结构特征和变异规律, 构建苜蓿属植物的系统发育关系, 结合上述苜蓿属植物来源地的生境特征, 探究我国苜蓿属物种的叶绿体基因组进化式样, 揭示我国苜蓿属植物的演化机理。
1 材料与方法1.1 植物材料
苜蓿属和胡卢巴属植物种子来自中国科学院昆明植物研究所中国西南野生生物种质资源库(Germplasm Bank of Wild Species, GBWS), 截形苜蓿种子来自澳大利亚南澳研究开发院(South Austrulian Research and Development Institute, SARDI)牧草种质资源库, 每种供试材料的采集编号以及采集地的年均降雨量、年均日照时数、年均气温和气候区特征见表 1。
表 1 取样的苜蓿属植物材料信息Table 1 Accession numbers of the Medicago materials used in this study
种属Taxon 编号
Accession # 采集地Locality 采集地点 年均降雨量/mm 年均日照时数/h 年均气温/℃ 气候区 单花胡卢巴
M. monantha GBWS 868710062997 新疆乌鲁木齐 286.2 2570 6.4 中温带大陆性干旱气候区 花苜蓿
M. ruthenica GBWS 868710187791 吉林松原 432.8 2879.8 4.5 中温带大陆性季风气候区 截形苜蓿
M. truncatula SARDI 南澳大利亚阿德莱德 365 2516 17.1 地中海气候区 毛荚苜蓿
M. edgeworthii GBWS 868710169173 西藏拉萨 426.4 3000 7.4 高原温带半干旱气候区 南苜蓿
M. polymorpha GBWS 868710272088 云南楚雄彝族自治州 864 2450 15.6 亚热带低纬高原季风气候区 青海苜蓿
M. archiducis-nicolai GBWS 868710200961 甘肃甘南藏族自治州 651.5 1800 5.1 大陆性季节气候区 天蓝苜蓿
M. lupulina GBWS 868710210600 上海市 1173.4 1885.9 17.6 亚热带季风性气候区 杂交苜蓿
M. varia GBWS 868710044970 新疆巴音郭楞蒙古自治州 58.6 3000 11.1 中温带和暖温带大陆性气候区 直果胡卢巴
M. orthoceras GBWS 868710063093 新疆乌鲁木齐 286.2 2570 6.4 中温带大陆性干旱气候区 紫苜蓿
M. sativa GBWS 868710125727 新疆巴音郭楞蒙古自治州 58.6 3000 11.1 中温带和暖温带大陆性气候区 胡卢巴
Trigonella sp. GBWS 868710059496 新疆塔什库尔干塔吉克自治县 68.3 4434.7 3.6 高原高寒干旱-半干旱气候区 GBWS, 中国科学院昆明植物研究所中国西南野生生物种质资源库;SARDI, 澳大利亚南澳研究开发院牧草种质资源库
1.2 温室培养
2018年至2019年, 表 1中苜蓿属和胡卢巴属植物的种子于智能温室(上海乾菲诺农业科技有限公司)进行萌发和培养。所有种子经温水浸种8 h后, 将种子置于25℃恒温箱中催芽, 每天投洗种子2次, 直至种子露白后, 进行播种。温室栽培条件为温度(25±2)℃, 光照强度400 μmol m-2 s-1, 空气湿度40%, 始终保持基质湿润。整个生育期内常规管理(图 1)。
 图 1 供试苜蓿属植物形态Fig. 1 Medicago plants cultivated in green house1, 紫苜蓿M. sativa;2, 杂交苜蓿M. varia;3, 截形苜蓿M. truncatula;4, 南苜蓿M. polymorpha;5, 天蓝苜蓿M. lupulina;6, 青海苜蓿M. archiducis-nicolai;7, 花苜蓿M. ruthenica;8, 直果胡卢巴M. orthoceras;9, 单花胡卢巴M. monantha;10, 毛荚苜蓿M. edgeworthii; 每个种各2张图片, 图示各物种的叶片、花序、荚果或复叶等
图 1 供试苜蓿属植物形态Fig. 1 Medicago plants cultivated in green house1, 紫苜蓿M. sativa;2, 杂交苜蓿M. varia;3, 截形苜蓿M. truncatula;4, 南苜蓿M. polymorpha;5, 天蓝苜蓿M. lupulina;6, 青海苜蓿M. archiducis-nicolai;7, 花苜蓿M. ruthenica;8, 直果胡卢巴M. orthoceras;9, 单花胡卢巴M. monantha;10, 毛荚苜蓿M. edgeworthii; 每个种各2张图片, 图示各物种的叶片、花序、荚果或复叶等1.3 DNA提取与高通量测序
采集幼嫩叶片立即置于液氮中冷冻后低温保存。样品总DNA采用mCTAB法提取, 具体步骤参考李金璐等[14]的植物DNA提取方法。
总DNA采用超声打断, 切胶回收350 bp的DNA片段, 利用NEBNext ©的建库试剂盒构建350 bp大小的文库, 利用Hiseq 4000 PE150的测序平台进行测序(北京诺禾致源科技股份有限公司, 北京)。
1.4 叶绿体基因组和ITS序列拼接与注释
高通量测序数据采用SPAdes3.6.1软件进行质量控制和拼接, 以已经发表的叶绿体基因组和ITS序列为参考, 用Blast程序筛选出叶绿体基因组和ITS序列的contigs[15], 筛选出的叶绿体基因组和ITS序列的contigs用Sequencher 4.10组装成完整的叶绿体基因组和ITS序列。
利用Plann程序对获得的叶绿体基因组进行注释。利用已经公开发表的M. sativa(序列接收号:MK460489)为参考序列, 对所有的蛋白质编码基因, rRNA和tRNA基因进行注释, 并对注释结果进行逐一检查。缺失的基因或者自动注释错误的基因, 依照参考序列的注释信息进行手动调整, 得到完整的注释结果。利用在线软件OGDRAW(https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)绘制苜蓿属物种的叶绿体基因组物理图谱。
1.5 简单重复序列分析
利用MISA软件对苜蓿属物种的叶绿体基因组的简单序列重复(simple sequence repeats, SSRs)分别进行检测。SSR位点的定义如下, 单核苷酸单元的重复数≥10;二核苷酸单元的重复数≥5, 三核苷酸单元重复数≥4, 四核苷酸、五核苷酸和六核苷酸单元的重复数≥3。两个SSRs之间的距离不少于20 bp。
1.6 系统发育分析
根据注释结果, 提取苜蓿属叶绿体基因组的76个编码基因, 利用MAFFT软件进行比对, 并利用MEGA X进行手工检查。比对好之后串联成一个数据集进行系统发育分析。用RAxML软件以最大似然法进行系统发育分析。核苷酸替换模型采用GTR+G, 系统发育各个分支长度的支持率通过进行500次自展重复分析获得。
2 结果与分析2.1 苜蓿属叶绿体基因组结构特征
本研究测定了苜蓿属10个物种完整的叶绿体基因组和1个胡卢巴属物种的叶绿体基因组, 平均测序深度从1, 222×(青海苜蓿)到10, 340×(花苜蓿)。供试材料的叶绿体基因组基本特征见表 2。叶绿体基因组大小不尽相同, 介于121—127 kb之间, 其中, 青海苜蓿叶绿体基因组最大, 长度为127, 358 bp, 单花胡卢巴最小, 长度为121, 082 bp, 两者基因组大小相差约6 kb。GC含量介于33.8%—34.1%之间, 平均值为34.02%, 不同物种间GC含量差异较小, 有5个物种的叶绿体GC含量完全一致, 均为34.1%。
表 2 苜蓿属植物叶绿体基因组测序分析Table 2 Sequencing and plastome statistics in Medicago plants
种属Taxon 测序数据量
Reads 平均深度
Depth 基因组大小/kb
Genome size GC含量/%
GC content 类型
Type 直果胡卢巴M. orthoceras 14, 111, 892 2665 121, 093 34.1 I 单花胡卢巴M. monantha 14, 248, 290 1773 121, 082 34.1 I 青海苜蓿M. archiducis-nicolai 14, 864, 700 1222 127, 358 34.2 II 毛荚苜蓿M. edgeworthii 12, 053, 011 2832 122, 598 33.9 I 花苜蓿M. ruthenica 27, 338, 408 10340 126, 792 34.1 II 紫苜蓿M. sativa 15, 254, 936 2620 125, 508 33.8 I 杂交苜蓿M. varia 17, 118, 231 2667 125, 364 33.8 I 南苜蓿M. polymorpha 13, 406, 640 3383 124, 349 34.1 I 截形苜蓿M. truncatula 12, 218, 607 2483 123, 973 34.0 I 天蓝苜蓿M. lupulina 13, 672, 134 3719 122, 242 34.1 III 胡卢巴Trigonella sp. 10, 206, 376 4210 125, 539 34.0 IV
由图 2可见, 供试苜蓿属和胡卢巴属均非典型的“四区”结构, 都缺失了一个大的反向重复序列, 属于豆科蝶形花亚科车轴草族反向重复序列缺失支(inverted repeat-lacking clade, IRLC)。依据组装注释的叶绿体基因组的结构特征, 供试材料的叶绿体基因组分为以下4种类型:1)类型I最为普遍, 包括图 2中所示的直果胡卢巴、单花胡卢巴、毛荚苜蓿、紫苜蓿、杂交苜蓿、南苜蓿和截形苜蓿;2)类型II, 包括青海苜蓿和花苜蓿两种;3)天蓝苜蓿单独作为类型III;4)胡卢巴单独作为类型IV。对比后发现, 四种类型的叶绿体基因组结构发生了不同程度的变化, 包括基因的重排现象、片段丢失等。
 图 2 苜蓿属植物4种叶绿体基因组类型Fig. 2 Four types of plastomes in Medicago plants最外圈的方框表示基因(圈内的表示顺时针转录, 圈外的表示逆时针转录);不同颜色表示不同功能的基因;阴影表示GC含量;类型I, 直果胡卢巴、单花胡卢巴、毛荚苜蓿、紫苜蓿、杂交苜蓿、南苜蓿和截形苜蓿;类型II, 青海苜蓿和花苜蓿;类型III, 天蓝苜蓿;类型IV, 胡卢巴
图 2 苜蓿属植物4种叶绿体基因组类型Fig. 2 Four types of plastomes in Medicago plants最外圈的方框表示基因(圈内的表示顺时针转录, 圈外的表示逆时针转录);不同颜色表示不同功能的基因;阴影表示GC含量;类型I, 直果胡卢巴、单花胡卢巴、毛荚苜蓿、紫苜蓿、杂交苜蓿、南苜蓿和截形苜蓿;类型II, 青海苜蓿和花苜蓿;类型III, 天蓝苜蓿;类型IV, 胡卢巴供试材料的叶绿体基因组都含有111个不同的基因, 包括77个蛋白编码基因、30个tRNA基因和4个rRNA基因。其中, 自我复制基因包括核糖体大亚基、核糖体小亚基的蛋白编码基因、tRNA和rRNA基因;光合作用基因包括光系统I、光系统II、NADH脱氢酶、细胞色素b/f复合物、ATP合成酶和RubisCo大亚基的蛋白编码基因;其他基因包括成熟酶K、囊膜蛋白、乙酰辅酶A羧化酶亚基、C-型细胞色素酶、蛋白酶基因以及未知功能蛋白编码基因(表 3)。
表 3 苜蓿属叶绿体基因组的基因类型、基因群和基因名称Table 3 Category, gene groups and genes identified in Medicago plastomes
类型Category 基因群
Gene groups 基因名称
Name of genes 自我复制 核糖体大亚基 rp133, rpl32, rpl20, rpl36, rpl14, rpl16, rpl2, rpl23 Self-replication 核糖体小亚基 rps15, rps4, rps14, rps2, rps18, rps12, rps11, rps8, rps3, rps19, rps7, rps12 DNA的RNA聚合酶 rpoB, rpoC1, rpoC2, rpoA 核糖体RNA基因 rrn16, rrn23, rrn4.5, rrn5 转运RNA基因 trnV-UAC, trnT-UGU, trnfM-CAU, trnS-UGA, trnE-UUC, trnY-GUA, trnD-GUC, trnS-GCU, trnQ-UUG, trnV-GAC, trnI-GAU, trnA-UGC, trnR-ACG, trnL-UAG, trnH-GUG, trnK-UUU, trnM-CAU, trnF-GAA, trnL-UAA, trnS-GGA, trnG-UCC, trnT-GGU, trnC-GCA, trnR-UCU, trnG-GCC, trnW-CCA, trnP-UGG, trnI-CAU, trnL-CAA, trnN-GUU 光合作用 光系统I psaC, psaA, psaB, psaI, psaJ Photosynthesis 光系统II psbM, psbB, psbT, psbH, psbA, psbZ, psbC, psbD, psbI, psbK, psbJ, psbL, psbF, psbE, psbN NADH脱氢酶 ndhH, ndhA, ndhI, ndhG, ndhE, ndhD, ndhF, ndhC, ndhK, ndhJ, ndhB 细胞色素b/f复合物 petA, petL, petG, petB, petD, petN ATP合成酶 atpB, atpE, atpI, atpH, atpF, atpA RubisCo大亚基 rbcL 其它基因 成熟酶K matK Other genes 囊膜蛋白 cemA 乙酰辅酶A羧化酶亚基 accD C-型细胞色素合成基因 ccsA 蛋白酶 clpP 未知蛋白功能基因 ycf1, ycf2, ycf3, ycf4
2.2 苜蓿属物种叶绿体基因组简单序列重复
由图 3可见, 苜蓿属叶绿体基因组的简单序列重复数为102到129个, 单碱基重复出现次数最多(62.14%), 其次为二碱基重复(21.29%)。单碱基重复多是A/T, 二碱基重复则多为AT/TA。
 图 3 苜蓿属物种叶绿体基因组简单重复序列的数量及其分布特征Fig. 3 Numbers and characterization of simple sequence repeat (SSR) in Medicago plastomes
图 3 苜蓿属物种叶绿体基因组简单重复序列的数量及其分布特征Fig. 3 Numbers and characterization of simple sequence repeat (SSR) in Medicago plastomes2.3 苜蓿属物种叶绿体基因组结构变异
如图 4所示, 在供试苜蓿属和胡卢巴属的叶绿体基因组中, 共发现10个倒位的结构变异:1)天蓝苜蓿中2个, 发生在基因atpB到ycf3、ndhC到trnLUAA之间的区域;2)青海苜蓿和花苜蓿共享4个倒位, 发生在从基因psbM到psaA、ndhB到trnNGUU、ndhB到rpoA和clpP到rpl20之间的区域;3)胡卢巴中6个, 发生在基因accD到trnPUGG、ndhB到rpoA、rpl20到psbF、ndhB到trnNGUU、psbJ到cemA以及psbB到ycf1之间的区域。其中, 胡卢巴发生倒位变异最多, 6处倒位既包括从psbB到ycf1共包含32个基因的长序列区段, 也有从psbJ到cemA仅包含3个基因的短序列区段。其中, 天蓝苜蓿独立发生2处倒位, 而在青海苜蓿、花苜蓿和胡卢巴3个物种中则平行共享以下2处倒位:ndhB到trnNGUU(包含12个基因)、ndhB到rpoA(包含14个基因)。
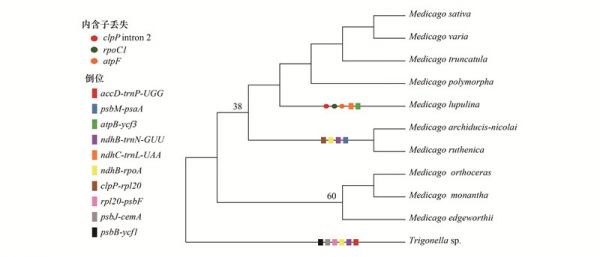 图 4 基于叶绿体编码基因的苜蓿属植物系统发育关系和叶绿体基因组结构变异Fig. 4 Phylogenetic relationships based on plastid coding genesand distribution of plastome structural mutations内含子丢失和倒位绘制在分支图的分枝上;右上角的小图表示带枝长的系统发育树, 图中数字表示所在节点的支持率, 未标注数字的节点的支持率均为100
图 4 基于叶绿体编码基因的苜蓿属植物系统发育关系和叶绿体基因组结构变异Fig. 4 Phylogenetic relationships based on plastid coding genesand distribution of plastome structural mutations内含子丢失和倒位绘制在分支图的分枝上;右上角的小图表示带枝长的系统发育树, 图中数字表示所在节点的支持率, 未标注数字的节点的支持率均为100叶绿体基因组序列分析还在天蓝苜蓿中发现了内含子丢失现象。3个发生内含子丢失的基因分别是clpP、rpoC1和atpF, 其功能分别为编码蛋白酶、DNA的RNA聚合酶以及ATP合成酶(图 4)。
2.4 基于叶绿体基因组序列和核ITS序列的苜蓿属系统发育关系
基于叶绿体基因的系统发育关系显示4个主要谱系分支, 从基部开始分别包含毛荚苜蓿、直果胡卢巴和单花胡卢巴、青海苜蓿和花苜蓿以及其他5个种。基于核基因ITS的系统发育关系与以上结果略有不同, 主要是天蓝苜蓿单独形成分支, 而不是与截形苜蓿等组成一个分支。ITS系统发育分化式样与传统的5个组的划分吻合。
3 讨论3.1 反向重复序列丢失和IRLC类群
作为分子生态学研究的基础, 分子测序技术的快速发展, 直接推动了叶绿体基因组学的发展[16]。典型陆生植物叶绿体基因组为双链环状DNA分子, 大小一般为115—165 kb, 常编码约101—118个基因。这些基因主要分为3类, 即光合作用有关基因、基因自身表达有关的基因和其他生物合成有关的基因。植物的叶绿体基因组结构一般具有相对保守的“四区”结构:大单拷贝区(large single copy region, LSC)、小单拷贝区(small single copy region, SSC)和两个序列完全相同、方向相反的反向重复区(inverted repeat region, IR)。在被子植物中, 反向重复区域的基因高度保守, 碱基的突变速率也低于单拷贝区, 研究表明IR区的存在对于稳定叶绿体基因组结构起到重要作用[17]。IR区主要编码4个rRNA基因(rrn16、rrn23、rrn4.5和rrn5)和一些未知功能基因(ycf)[18]。被子植物叶绿体基因组的“四区”结构相对保守, 但IR区域可扩张或缩减, 是影响叶绿体基因组大小的重要因素之一, 导致不同植物类群的叶绿体基因组差异很大[19]。例如, 发生在豌豆(Pisum sativum)中IR完全删减[20]和天竺葵(Pelargonium hortorum)中IR极度增加(增至76 kb)[21]的极端报道。此外, 已报道的樟科叶绿体基因片段丢失事件, 导致该科植物叶绿体基因组整体缩小, 并且和樟科种系发生进程一致[22]。
物种的进化必然是建立在基因组的进化之上, 物种多样性是生态、进化和历史过程的产物, 其演化路径可能既有宏观上的方向性, 也有微观成种的随机性。叶绿体基因组的缩减、扩张及丢失可能与物种在进化过程中的物种分化和谱系分化相吻合, 当然, 两者也不一定完全等同[23]。目前, 尽管对植物叶绿体基因组的结构和功能有所认识, 但有关叶绿体基因组结构变异与其生态进化起源、演化驱动机制等尚待深入研究。自Kolodner & Tewari[24]首次在豌豆叶绿体基因组中发现IR丢失现象, 之后的研究又陆续在绿豆(Vigna radiata)、大豆(Glycine max)、菜豆(Phaseolus vulgaris)和蚕豆(Vicia faba)中证实了其叶绿体基因组IR缺失现象[25—26]。随着叶绿体基因组数据的不断积累, 豆科蝶形花亚科车轴草族物种的叶绿体基因组发生IR区的丢失现象, 成为该类植物的指示特征, 为此, 该类群也被称为反向重复序列缺失支, 并且IR丢失事件(该支的分化事件)发生在—3900万年前[27—30]。如图 2所示, 供试苜蓿属及其近缘属胡卢巴也呈现IR区域的丢失, 均为IRLC类成员(图 2)。越来越多的证据发现, IR区段丢失现象还发生在其它植物类群中, 如牻牛儿苗科(Geraniaceae)的老鹳草属(Geranium)[31]和仙人掌科(Cactaceae)的仙人掌(Carnegiea gigantea)[32]中, 进一步表明IR区的丢失可能在被子植物谱系中多次独立发生, 而叶绿体基因组大的结构变异在特定类群因具有强烈的系统发育信号, 常被用来定义单系类群[33]。
3.2 苜蓿属叶绿体基因组结构变异的进化启示
包括苜蓿属在内的豆科IRLC类群是IR收缩的经典案例, 目前认为大的IR收缩可能与基因组的异常重组有关[34]。研究发现, 小苜蓿(M. minima)中一段新的IR位于ycf1和clpP附近, 而这两个基因序列通常被认为是高度变化的, 并可能是基因重组的热点区域[23]。碱基替换和结构变异引起的基因重组事件频发也可能导致豆科植物IR收缩的发生[35]。叶绿体基因组的大小除了受IR扩张和收缩的影响, 还与重复序列的含量相关。本研究测定的我国苜蓿属植物的叶绿体基因组简单序列重复数为102到129个, 单碱基重复是出现次数最多的(62.14%), 可作为分子标记, 为研究苜蓿系统学和进化生物学提供依据。然而, 叶绿体基因组的稳定性和重复序列累积以及IR缺失或是获得之间的关系尚未明了, 特别是IR扩张、收缩或缺失等结构变异对核酸替换率的影响尚不清晰。
除反向重复序列(IR)的缺失, 供试苜蓿属物种中还发现了其它显著的叶绿体基因组结构变异, 如天蓝苜蓿clpP、rpoC1和atpF基因的内含子丢失、青海苜蓿和花苜蓿之间共享倒位等, 这些独立或是平行发生的结构变异, 也是潜在的系统发育信号, 可以作为区分不同类群的重要特征(图 4)。研究发现, 豆科菜豆亚族的刺槐属(Robinia)、绿豆、菜豆等物种, 其叶绿体基因组发生50 kb倒位, 和其它具有该结构变异的类群构成了50 kb倒位支[36—38]。事实上, 包括苜蓿属在内的核心车轴草族的叶绿体基因组也发生了不同程度的重排, 包括基因的重复、丢失和序列的倒位等[39]。大于1kb的倒位在蝶形花亚科植物中较为常见, 本研究中青海苜蓿和花苜蓿倒位发生在基因psbM到psaA、ndhB到trnNGUU、ndhB到rpoA以及clpP到rpl20之间, 而近缘属的胡卢巴倒位变异则高达6处, 包括psbB到ycf1的区段(含32个基因), 以及psbJ到cemA区段(含3个基因), 显示出叶绿体基因组结构变异的多样性和复杂性。
本研究结果发现, 我国天蓝苜蓿中的内含子丢失与其它研究报道相一致, 有必要通过更加广泛的取样和分析, 进一步评估该分支苜蓿物种的倒位、内含子丢失及其演化模式[23]。研究表明, 反向重复序列可能通过介导分子内重组而导致倒位的发生[28]。缺乏反向重复区的豆科蝶形花亚科IRLC物种如何控制叶绿体基因组稳定性还远未被阐明, 或许对苜蓿属广泛的叶绿体基因组分析可以找出结构稳定性与多样性的关系, 从而揭示IR多次得失的变异机制。
3.3 我国苜蓿属植物的演化路径
我国野生苜蓿分布在全国不同地理区域, 适应各种生境特征, 从寒冷的北部沙漠到温暖潮湿的南部山区和中部平原地带, 从国内东部沿海到喜马拉雅山脉[2]。这些野生种拥有丰富的自然变异来源, 是了解苜蓿属植物种群动态和改善苜蓿品种的重要基础。对于植物的生长和分布来说, 温度、降水和光照是最重要的气候因子, 强烈地影响着物种的分布格局(表 1)。由本实验测序结果(图 4), 对照表 1生境特征可知, 苜蓿属植物的进化与年平均温度、年均降雨量、年均日照时数等生境因子之间存在密切关系, 特别是年平均温度影响最大。年均温度越高, 物种出现的时间越晚, 进化程度越高, 如紫苜蓿(11.1℃)、截形苜蓿(17.1℃)、南苜蓿(15.6℃)、天蓝苜蓿(17.6℃), 反之, 处于进化分支树基部的物种, 其生境条件均相对较为恶劣, 如胡卢巴(3.6℃)、毛荚苜蓿(7.4℃)、单花胡卢巴(6.4℃)、花苜蓿(4.5℃)、青海苜蓿(5.1℃), 表明温度是苜蓿属植物物种进化的重要影响因子, 温度越高, 越利于苜蓿属植物的生长, 有利于其拓展更广阔的生境, 进而成为广布种。其中, 天蓝苜蓿是一年生、二年生或偶尔短命的多年生植物, 自交为主(自交率大于95%), 分布广泛, 是易于繁衍的绿肥植物;而花苜蓿则是长寿的多年生植物, 行异交(自交率低于30%), 分布狭窄, 适应干燥极寒的砂砾和沙漠等低投入生态系统。此外, 天蓝苜蓿具有不裂的小豆荚, 通过生物和非生物因子促进种子的长距离传播, 而花苜蓿具开裂的豆荚并缺乏有效的种子传播机制。从表型上看, 天蓝苜蓿的花相对较小, 具2—4 mm的黄色蝶形花冠, 而花苜蓿则有长达8 mm的黄色花冠, 更大更艳丽, 能够吸引更多的昆虫访花, 如蜜蜂、大黄蜂等。然而, 随着快速城市化和过度放牧, 国内的野生苜蓿资源受到人为干扰和城市化进程的影响, 种群数量和规模大幅减少, 其野生种质资源调查和保护需引起重视。
苜蓿属和胡卢巴属植物具有相似的特征, 长期以来, 二者之间的进化分类界限不明显, 部分物种的归属存在争议, 两个属之间存在“类苜蓿植物(Medicagoid)”或“类胡卢巴植物(Trigonelloid)”[40]。邓宏超等[4]开展了苜蓿属和胡卢巴属植物形态特征及数量分类研究发现, 直果胡卢巴和单花胡卢巴属于角形果组, 聚类结果显示它们与苜蓿属植物之间存在较近的亲缘关系, 建议进一步开展深入研究确定其能否归并到苜蓿属。而早期Small等[41]也曾提出将胡卢巴归至角形果组并合并到苜蓿属中。本研究基于叶绿体基因组及ITS序列重新构建了苜蓿属系统发育关系, 我国苜蓿属物种的基因分化式样与传统分类的紫苜蓿组、南苜蓿组、阔荚苜蓿组和天蓝苜蓿组基本吻合。其中, 直果胡卢巴和单花胡卢巴与毛荚苜蓿形成一枝, 分子证据支持将苜蓿属和胡卢巴属之间的过渡类型“类苜蓿植物”独立出来(图 4和图 5)。由此可见, 叶绿体基因组大量的系统发育信息位点, 不仅有助于阐明苜蓿属和胡卢巴属之间的系统发育关系, 而且可建立高分辨率的苜蓿属和胡卢巴属种间的生态进化关系。生命形式、交配系统和种子传播是影响苜蓿属植物种群的遗传结构塑造和地理分布的适应性性状。以上苜蓿属植物叶绿体和核基因组分析连同生态、表型数据可为该属植物的演化路径提供依据, 并有助于确定自然种群的进化潜力和保护策略。
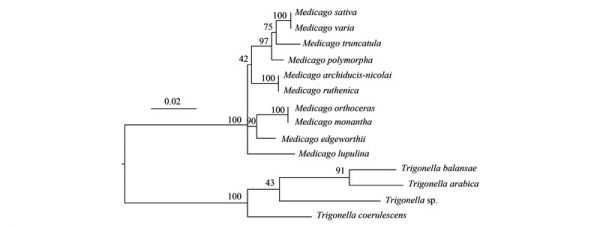 图 5 基于rDNA的ITS序列的苜蓿属植物系统发育树Fig. 5 Phylogenetic tree of Medicago species based on rDNA ITS sequences
图 5 基于rDNA的ITS序列的苜蓿属植物系统发育树Fig. 5 Phylogenetic tree of Medicago species based on rDNA ITS sequences 参考文献
[1]Small E, Jomphe M. A synopsis of the genus Medicago (Leguminosae). Canadian Journal of Botany, 1989, 67: 1604-1606. DOI:10.1139/b89-201
[2]Small E. Alfalfa and relatives: evolution and classification of Medicago. Ottawa: NRC Research Press, 2011: 727.
[3]中国科学院中国植物志编辑委员会. Flora of China. Medicago, 2010, 10: 553-557.
[4]邓宏超, 崔大方, 羊海军, 李飞飞, 方颖. 苜蓿属和胡卢巴属植物的形态特征及数量分类研究. 植物资源与环境学报, 2010, 19(4): 1-11.
[5]Maureira-Butler I J, Pfeil B E, Muangprom A, Osborn T C, Doyle J J. The reticulate history of Medicago (Fabaceae). Systematic Biology, 2008, 57(3): 466-482. DOI:10.1080/10635150802172168
[6]Ivanov, A I. History, origin and evolution of the genus Medicago, subgenus Falcago. Bulletin of Applied Genetics and Plant-breeding, 1977, 59: 3-40.
[7]Li X H, Wang X J, Wei Y L, Brummer E C. Prevalence of segregation distortion in diploid alfalfa and its implications for genetics and breeding applications. Theoretical and Applied Genetics, 2011, 123(4): 667-679. DOI:10.1007/s00122-011-1617-5
[8]Rose R J. Medicago truncatula as a model for understanding plant interactions with other organisms, plant development and stress biology: past, present and future. Functional Plant Biology, 2008, 35: 253-264. DOI:10.1071/FP07297
[9]Shinozaki K, Ohme M, Tanaka M, Wakasugi T, Hayashida N, Matsubayashi T, Zaita N, Chunwongse J, Obokata J, Yamaguchi-Shinozaki K. The complete nucleotide sequence of the tobacco chloroplast genome its gene organization and expression. EMBO Journal, 1986, 5(9): 2043-2049. DOI:10.1002/j.1460-2075.1986.tb04464.x
[10]Ohyama K, Fukuzawa H, Kohchi T, Shirai H, Sano T, Sano S, Umesono K, Shiki Y, Takeuchi M, Chang Z, Aota S, Inokuchi H, Ozeki H. Chloroplast gene organization deduced from complete sequence of liverwort Marchantia polymorpha chloroplast DNA. Nature, 1986, 322(6079): 572-574. DOI:10.1038/322572a0
[11]Wang W C, Chen S Y, Guo W, Li Y Q, Zhang X. Tropical plants evolve faster than their temperate relatives: a case from the bamboos (Poaceae: Bambusoideae) based on chloroplast genome data. Biotechnology & Biotechnological Equipment, 2020, 34(1): 482-493.
[12]Song Y, Yu W B, Tan Y H, Liu B, Yao X, Jin J J, Padmanaba M, Yang J B, Corlett R T. Evolutionary comparisons of the chloroplast genome in Lauraceae and insights into loss events in the Magnoliids. Genome Biology and Evolution, 2017, 9(9): 2354-2364. DOI:10.1093/gbe/evx180
[13]Shaver J M, Oldenburg D J, Bendich A J. Changes in chloroplast DNA during development in tobacco, Medicago truncatula, pea, andmaize. Planta, 2006, 224(1): 72-82. DOI:10.1007/s00425-005-0195-7
[14]李金璐, 王硕, 于婧, 王玲, 周世良. 一种改良的植物DNA提取方法. 植物学报, 2013, 1: 72-78.
[15]Altschul S F, Madden T L, Schäffer A A, Zhang J, Zhang Z, Miller W, Lipman D J. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Research, 1997, 25: 3389-3402. DOI:10.1093/nar/25.17.3389
[16]Bock R. Plastid biotechnology: prospects for herbicide and insectresistance, metabolic engineering and molecular farming. Current Opinion in Biotechnology, 2007, 18(2): 100-106. DOI:10.1016/j.copbio.2006.12.001
[17]Jansen R K, Ruhlman T A. Plastid genomes of seed plants. In: Bock R, Knoop V. Genomics of chloroplasts and mitochondria. Dordrecht: Springer Netherlands, 2012, 103-126.
[18]Gao L, Su Y J, Wang T. Plastid genome sequencing, comparative genomics, and phylogenomics: current status and prospects. Journal of Systematics and Evolution, 2010, 48(2): 77-93. DOI:10.1111/j.1759-6831.2010.00071.x
[19]Mower J P, Vickrey T L. Structural diversity amongplastid genomes of land plants. In: Chaw SM, Jansen RK. Advances in botanical research. London UK: Academic Press, 2018, 263-292
[20]Saski C, Lee S B, Daniell H, Wood T C, Tomkins J, Kim H G, Jansen R K. Complete chloroplast genome sequence of Glycine max and comparative analysis with other legume genomes. Plant Molecular Biolology, 2005, 59(2): 309-322. DOI:10.1007/s11103-005-8882-0
[21]Chumley T W, Palmer J D, Mower J P, Fourcade H M, Calie P J, Boore J L, Jansen R K. The complete chloroplast genome sequence of Pelargonium x hortorum: organization and evolution of the largest and most highly rearranged chloroplast genome of land plants. Molecular Biology and Evolution, 2006, 1: 2175-2190.
[22]Song Y, Yu W B, Tan Y H, Liu B, Yao X, Jin J J, Padmanaba M, Yang J B, Corlett R T. Evolutionary comparisons of the chloroplast genome in Lauraceae and insights into loss events in the Magnoliids. Genome Biology and Evolution, 2017, 9(9): 2354-2364. DOI:10.1093/gbe/evx180
[23]Choi I S, Jansen R, Ruhlman T. Lost and found: return of the inverted repeat in the legume clade defined by its absence. Genome Biology and Evolution, 2019, 11(4): 1321-1333. DOI:10.1093/gbe/evz076
[24]Kolodner R, Tewari K K. Inverted repeats in chloroplast DNA fromhigher plants. Proceedings of the National Academy of Sciences of the United States of America, 1979, 76(1): 41-45. DOI:10.1073/pnas.76.1.41
[25]Palmer J D, Thompson W F. Rearrangements in the chloroplast genomes of mung bean and pea. Proceedings of the National Academy of Sciences of the United States of America, 1981, 78(9): 5533-5537. DOI:10.1073/pnas.78.9.5533
[26]Palmer J D, Singh G P, Pillay D. Structure and sequence evolution of three legume chloroplast DNAs. Molecular and General Genetics, 1983, 190(1): 13-19. DOI:10.1007/BF00330318
[27]Palmer J D, Osorio B, Aldrich J, Thompson W F. Chloroplast DNA evolution among legumes: loss of a large inverted repeat occurred prior to other sequence rearrangements. Current Genetics, 1987, 11(4): 275-286. DOI:10.1007/BF00355401
[28]Lavin M, Doyle J J, Palmer J D. Evolutionary significance of the loss of the chloroplast-DNA inverted repeat in the Leguminosae subfamily Papilionoideae. Evolution, 1990, 44(2): 390-402.
[29]McMahon M M, Sanderson M J. Phylogenetic supermatrix analysis of GenBank sequences from 2228 papilionoid legumes. Systematic Biology, 2006, 55(5): 818-836. DOI:10.1080/10635150600999150
[30]Schwarz E N, Ruhlman T A, Weng M L, Khiyami M A, Sabir J S M, Hajarah N H, Alharbi N S, Rabah S O, Jansen R K. Plastome-wide nucleotide substitution rates reveal accelerated rates in Papilionoideae and correlations with genome features across legume subfamilies. Journal of Molecular Evolution, 2017, 84(4): 187-203. DOI:10.1007/s00239-017-9792-x
[31]Guisinger M M, Kuehl J V, Boore J L, Jansen R K. Extreme reconfiguration of plastid genomes in the angiosperm family Geraniaceae: rearrangements, repeats, and codon usage. Molecular Biology and Evolution, 2011, 28(1): 583-600. DOI:10.1093/molbev/msq229
[32]Sanderson M J, Copetti D, Búrquez A, Bustamante E, Charboneau J L M, Eguiarte L E, Kumar S, Lee H O, Lee J, Mcmahon M. Exceptional reduction of the plastid genome of saguaro cactus (Carnegiea gigantea): loss of the ndh gene suite and inverted repeat. American Journal of Botany, 2015, 102(7): 1115-1127. DOI:10.3732/ajb.1500184
[33]Lavin M, Herendeen P S, Wojciechowski M F. Evolutionary rates analysis of Leguminosae implicates a rapid diversification of lineages during the Tertiary. Systems Biology, 2005, 54(4): 575-594. DOI:10.1080/10635150590947131
[34]Blazier J C, Jansen R K, Mower J P, Govindu M, Zhang J, Weng M L, Ruhlman T A. Variable presence of the inverted repeat and plastomestability in Erodium. Annals of Botany, 2016, 114: 1209-1220.
[35]Magee A M, Aspinall S, Rice D W, Cusack B P, Semon M, Perry A S, Stefanovic' S, Milbourne D, Barth S, Palmer J D, Gray J C, Kavanagh T A, Wolfe K H. Localized hypermutation and associated gene losses in legume chloroplast genomes. Genome Research, 2010, 20(12): 1700-1710. DOI:10.1101/gr.111955.110
[36]Palmer J D, Osorio B, Thompson W F. Evolutionary significance of inversions in legume chloroplast DNAs. Current Genetics, 1988, 14(1): 65-74. DOI:10.1007/BF00405856
[37]Doyle J J, Doyle J L, Ballender J A, Palmer J D. The distribution and phylogenetic significance of a 50-kb chloroplast DNA inversion in the flowering plant family Leguminosae. Molecular Phyogenetics and Evolution, 1996, 5(2): 429-438. DOI:10.1006/mpev.1996.0038
[38]Schwarz E N, Ruhlman T A, Sabir J S M. Plastid genome sequences of legumes reveal parallel inversions and multiple losses of rps16 in Papilionoid. Journal of Systematics and Evolution, 2015, 53(5): 458-468. DOI:10.1111/jse.12179
[39]Cai Z Q, Guisinger M, Kim H G, Ruck E, Blazier J C, Mcmurtry V, Kuehl J V, Boore J, Jansen R K. Extensive reorganization of the plastid genome of Trifolium subterraneum (Fabaceae) is associated with numerous repeated sequences and novel DNA insertions. Journal of Molecular Evolution, 2008, 67(6): 697-704.
[40]Small E. A taxonomic study of the "medicagoid" Trigonella (Leguminosae). Canadian Journal of Botany, 1987, 65: 1199-1211. DOI:10.1139/b87-166
[41]Small E, Cranpton C W, Brooks B S. The taxonomic value of floral characters in tribe Trigonelleae (Leguminosae), with special reference to Medicago. Canadian Journal of Botany, 1981, 59(9): 1578-1598. DOI:10.1139/b81-216
相关知识
胡光万
中国科学院昆明植物研究所导师教师师资介绍简介
刘红梅
顾翠花
高连明
Genetic Structure Analysis of Different Populations of Spotted Sea Bass (Lateolabrax maculatus)
廖晨阳
中国木犀科(Oleaceae)植物花粉形态研究及其系统演化的探讨(Ⅱ)—木犀族(Oleeae)和茉莉属(Jasminum)的花粉形态研究
野生牡丹种质资源现状及保护建议
孔宏智
网址: The Evolution Path of Medicago in China based on the Chloroplast Genome Analysis https://m.huajiangbk.com/newsview415228.html
| 上一篇: 品一品宋人以花为中心,写花、赏花 |
下一篇: 科学家解析猴面包树属植物的演化历 |